Dr. Anna Heintz-Buschart
Kontakt / Adresse
Dr. Anna Heintz-Buschart
ehemalige Wissenschaftliche Mitarbeiterin
Department Bodenökologie
Helmholtz-Zentrum für Umweltforschung - UFZ
Theodor-Lieser-Str. 4 | 06120 Halle/Saale
Metagenomics Support Unit
Deutsches Zentrum für integrative Biodiversitätsforschung (iDiv) Halle-Jena-Leipzig
Email:
Twitter:


Forschungsinteressen am UFZ und iDiv (2017-2021)
Seit August 2021 arbeite ich nicht mehr am UFZ und iDiv. Für Fragen zum Metagenomics Support nehmen Sie bitte Kontakt mit
Prof. Dr. François Buscot
auf.
“Omics” und bioinformatische Methoden für die integrative Analyse mikrobieller Gemeinschaften
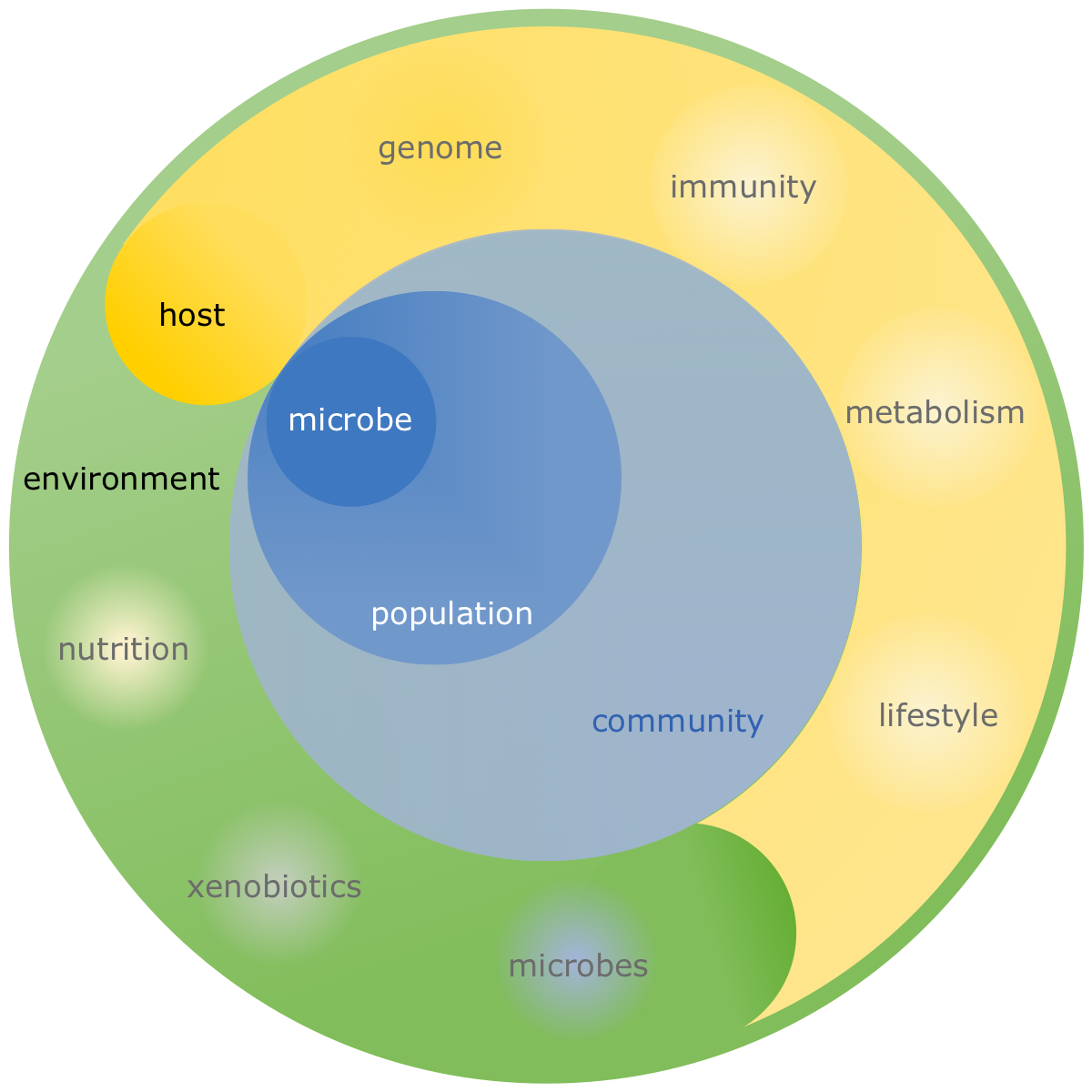 Die Genome von Mikroorganismen in Gemeinschaften werden durch die Wechselbeziehungen mit Verwandten (der selben Population), mit anderen Mikroorganismen und mit der Umwelt geformt. Dazu gehören auch Wechselbeziehungen zu mehrzelligen Wirtsorganismen, deren Genome, Immunsysteme, Stoffwechsel und Lebenstile besitmmen, welchen Nährstoffen, potenziell anti-mikrobiellen Fremdstoffen und anderen Mikroorganismen die mikrobielle Gemeinschaft ausgesetzt ist.
Die Genome von Mikroorganismen in Gemeinschaften werden durch die Wechselbeziehungen mit Verwandten (der selben Population), mit anderen Mikroorganismen und mit der Umwelt geformt. Dazu gehören auch Wechselbeziehungen zu mehrzelligen Wirtsorganismen, deren Genome, Immunsysteme, Stoffwechsel und Lebenstile besitmmen, welchen Nährstoffen, potenziell anti-mikrobiellen Fremdstoffen und anderen Mikroorganismen die mikrobielle Gemeinschaft ausgesetzt ist.
Mikrobielle Gemeinschaften spielen eine wichtige Rolle in Stoffkreisläufen und Energieflüssen, und sie gehen enge Wechselbeziehungen mit Makroorganismen ein. Diese Verbindungen sind von grundlegender Wichtigkeit für die meisten natürlichen und landwirtschaftlichen Ökosysteme und auch für die menschliche Gesundheit. Allerdings ist über die Abläufe, die zum Zusammenkommen und Erhalt dieser Interaktionen führen, bislang vieles nicht bekannt. Modernste Meta-“omics” Technologien, wie die Metagenomik, Metatranskriptomik und Metaproteomik können Fragen an mikrobiellen Gemeinschaften beantworten.
welche Mikroorganismen sind präsent? was ist ihr funktionelles Potenzial, das heißt, welche Gene besitzen ihre Genome? welche dieser Gene werden exprimiert - in der ganzen mikrobiellen Gemeinschaft oder von spezifischen Mitgliedern, und wenn ja von welchen? welche Enzyme, welche Stoffwechselprodukte, und was für Proteinstrukturen sind zu finden? das heißt, was tun die Mikroben gerade? Die Antworten auf diese Fragen tragen zu tieferen Einsichten in die Fähigkeiten und Wechselwirkungen innerhalb der mikrobiellen Gemeinschaften und auch in deren Einfluss auf Wirtsorganismen bei. Allerdings sind all diese Antworten “versteckt” in großen Datensätzen. Ich beschäftige mich damit, biologische Interpretationen zu ermöglichen und verwende dafür bioinformatische Ansätze. Diese Ansätze werden für jedes Projekt angepasst oder entwickelt, sodass die jeweils bedeutenden Daten extrahiert werden.
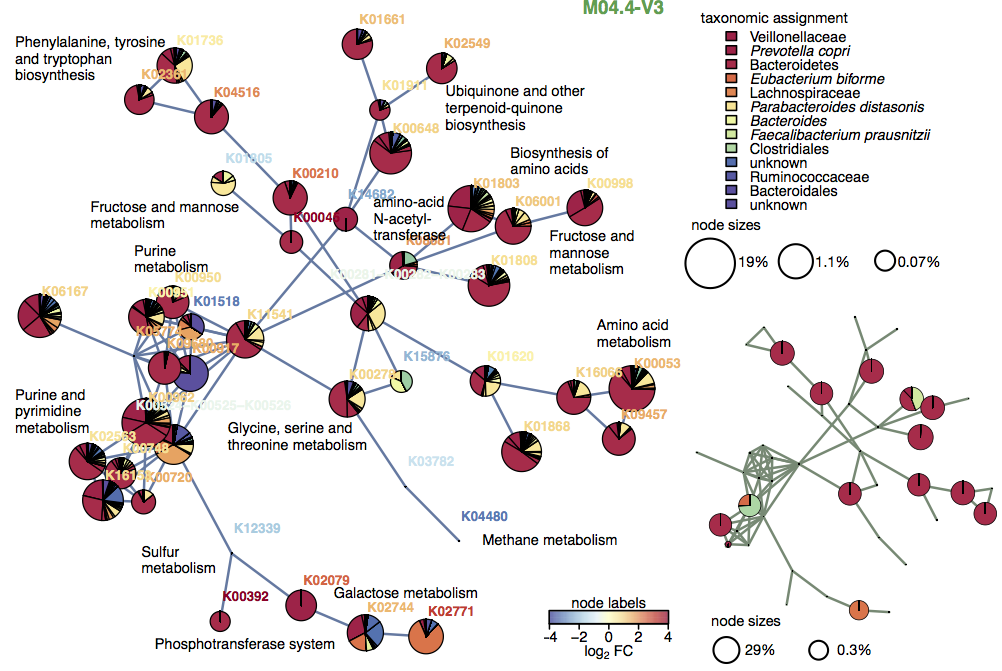
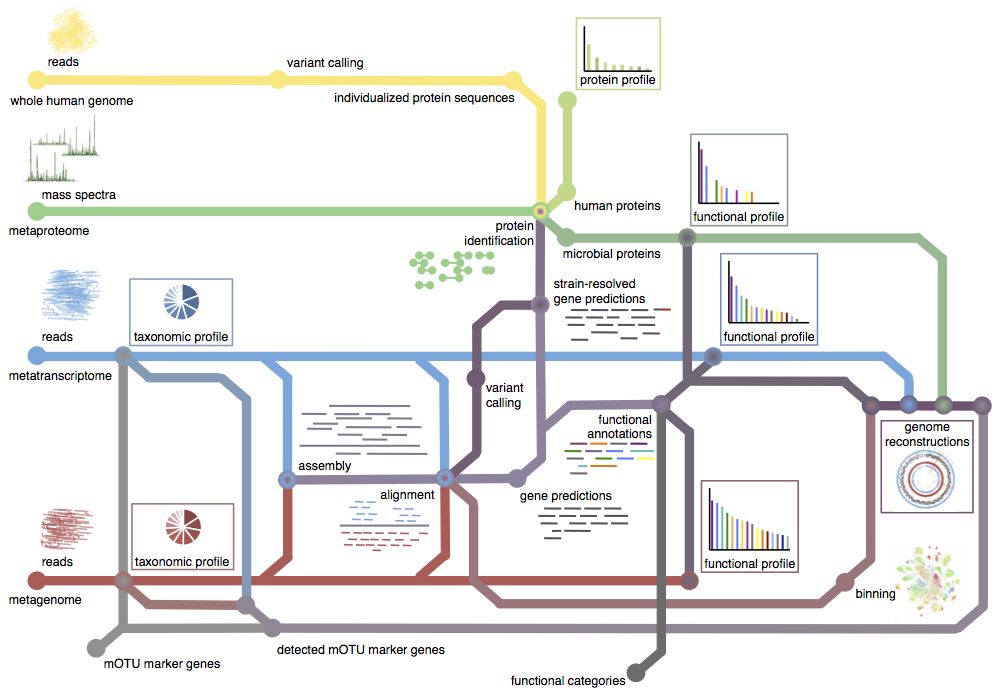
Neben der Prozessierung verschiedener “omics” Datensätze ist die Integration verschiedener Ebenen wichtig. Dafür werden Datenbanken sowie statistische Methoden benötigt. Andererseits ergeben sich aus multiplen “omics” Ebenen Synergien, da Informationen von einer Ebene zur besseren Messbarkeit oder Interpretation auf anderen Ebenen beitragen. Schließlich können durch bioinformatische Methoden Kontexte und Bezüge hergestellt werden, die zusammen mit Visualisierungen bei der Beantwortung biologischer Fragestellungen helfen.
Forschungsprojekte (2017-2021)
Jena Experiment
Wir untersuchen die Rolle von arbuskulärer Mykorrhiza in der Etablierung langfristiger Biodiversität-Ökosystemfunktions Beziehungen im Jena Experiment. Das Projekt, das ich mit François Buscot anleite, ist im Januar 2020 mit Cynthia Albracht als Doktorandin gestartet. In dem verwandten iDiv Flexpool Projekt RESFun kooperieren wir mit Prof. Alexandra Weigelt von der Uni Leipzig und ihrem Doktoranden Justus Hennecke. Sie untersuchen, welche Pilzpartner Pflanzen anziehen, deren Wurzeln unterschiedliche Strategien für die Erschließung von Bodenresourcen haben.
DiscoMole und iSBio
Durch DFG-Finanzierung im Rahmen der NGS calls konnte unser iDiv Konsortium "Soil Biodiversity and Function" Metagenome aus Bodenproben der iSBio-Initiative sequenzieren. Spannend an dieser Sammlung ist, dass neben der genetischen Information weitreichende funktionale Informationen für die gleichen Proben aufgenommen wurden. Damit möchten wir insbesondere das Verhältnis von mikrobieller Diversität und diesen Bodenfunktionen ausloten. Im Rahmen eines iDiv Flexpool Projekts erstellt Dr Qicheng Bei Szenarien-basierte Modelle der Entwicklung von pathogenen Organismen.
Weitere Kollaborationen:
Durch mein Engagement für die Omics und Bioinformatics Plattform von 2017-2021 in der Bodenökologieabteilung arbeitete ich mit den meisten Kollegen in dieser Abteilung zusammen. Daneben bestanden Projekte mit Wissenschaftlern vom iDiv.
Die "global change experimental facility" (GCEF) ist ein Feldexperiment, in dem Konsequenzen des Klimawandels unter verschiedenen Landnutzungstypen simuliert werden. Mit Claudia Breitkreutz, Marie-Lara Bouffaud, Mika Tarkka und Thomas Reitz untersuchten wir, wie sich mikrobielle Gemeinschaften und mikrobielle Prozesse verändern. Mit Witoon Purahong und Sara Wahdan untersuchten wir die Zersetzung von Pflanzenmaterial und Kunststoffen. Dabei verwendeten wir Omics, um die Aktivität der Bakterien und Pilze, die an der Zersetzung beteiligt sind, genauer zu beschreiben.
Die Biodiversitätsexploratorien sind ein Kernprojekt unserer Abteilung. Julia Moll untersucht Bakterien, Archäen und Pilze, die mikrobielle Gemeinschaften und Netzwerke in Totholz bilden. Mit Kezia Goldmann haben wir eine Studie über die räumliche und zeitliche Diversität von arbuskulären Mykorrhizapilzen in einem Grasland abgeschlossen: Scalemic.
Im SPP2089 Rhizosphere Spatiotemporal Organisation untersuchen Mika Tarkka und Minh Ganther die Genexpression in räumlich und zeitlich aufgelösten Experimenten mit Maiswurzeln. Sie werden in den nächsten Jahren auch Pflanzen-Mikroorganismen-Interaktionen miteinbeziehen.
Die Arbeit von François Buscots Doktoranden Luis Daniel Prada Salcedo und Jean de Dieu Habiyaremye über Bodenmikrobiota im Soil for Europe Projekt und in den PhytOakmetern von Sylvie Herrmann wurde durch Pipeline-Entwicklung unterstützt.
Wissenschaftler und Projekte vom iDiv, die Böden von unterschiedlichen Perspektiven untersuchen, haben sich im "Soil Biodiversity and Function" Projekt zusammen geschlossen um die Biodiversität im Boden besser zu verstehen und Bodenökologie in der Biologenausbildung zu stärken.
In dem iDiv Soil Biodiversity and Function Konsortium erkunden wir mikrobielle Gemeinschaften und Prozesse über Umweltgradienten mit Nico Eisenhauers Experimental Interaction Ecology Gruppe und Ika Djukics TeaComposition initiative.
Kezia Goldmann und ich haben mit Olga Ferlian aus Nico Eisenhauers Experimental Interaction Ecology Gruppe die Zusammensetzung und Vielfalt von Mykorrhizapilzen im myDiv Experiment bestimmt.
Ich arbeite zusammen mit Hjalmar Kühl und anderen vom Max Planck Institute for Evolutionary Anthropology und Alexandra Muellner-Riehls Molecular Evolution and Plant Systematics Gruppe an der Universität Leipzig am Pan-African Chimpanzee Microbiome and Diet Research Project.
Mit der Arbeitsgruppe von Marie Spohn, vormals Boden Biogeochemie an der Universität Bayreuth, haben wir die Konsequenzen langfristiger Stickstoff- und Phosphatdüngung auf mikrobielle Gemeinschaften im Graslandboden.
Ausgewählte Publikationen
M. Marselle, T. Hartig, D. Cox, S. de Bell, S. Knapp, S. Lindley, M. Triguero-Mas, K. Böhning-Gaese, P.A. Cook, S. de Vries, A. Heintz-Buschart, M. Hofmann, K.N. Irvine, N. Kabisch, F. Kolek, R. Kraemer, I. Markevych, D. Martens, R. Müller, M. Nieuwenhuijsen, J.M. Potts, J. Stadler, S. Walton, S.L. Warber, A. Bonn (2021): Pathways linking biodiversity to human health: a conceptual framework. Environment International, 150: 106420.
C. Weißbecker, B. Schnabel, A. Heintz-Buschart (2020): dadasnake, a Snakemake implementation of DADA2 to process amplicon sequencing data for microbial ecology. GigaScience 9: giaa135.
C.A. Guerra, A. Heintz-Buschart, J. Sikorski, A. Chatzinotas, N. Guerrero-Ramírez, S. Cesarz, L. Beaumelle, M.C. Rillig, F.T. Maestre, M. Delgado-Baquerizo, F. Buscot, J. Overmann, G. Patoine, H.R.P. Phillips, M. Winter, T. Wubet, K. Küsel, R.D. Bardgett, E.K. Cameron, D. Cowan, T. Grebenc, C. Marín, A. Orgiazzi, B.K. Singh, D.H. Wall, N. Eisenhauer (2020): Blind spots in global soil biodiversity and ecosystem function research. Nature Communications 11: 3870.
P.-M. Schleuss, M. Widdig, A. Heintz-Buschart, A. Guhr, S. Martin, K. Kirkman, M. Spohn (2019): Stoichiometric controls of soil carbon and nitrogen cycling after long-term nitrogen and phosphorus addition in a mesic grassland in South Africa. Soil Biology & Biochemistry 135: 294-303.
L. Wampach*, A. Heintz-Buschart*, J.V. Fritz*, J. Ramiro-Garcia, J. Habier, M. Herold, S. Narayanasamy, A. Kaysen, A.H. Hogan, L. Bindl, J. Bottu, R. Halder, C. Sjöqvist, P. May, A.F. Andersson, C. de Beaufort, P. Wilmes (2018): Birth mode is associated with earliest strain-conferred gut microbiome functions and immunostimulatory potential. Nature Communications 9: 5091.
A. Heintz-Buschart, P. May, C.C. Laczny, L.A. Lebrun, C. Bellora, A. Krishna, L. Wampach, J.G. Schneider, A. Hogan, C. de Beaufort, P. Wilmes (2016): Integrated multi-omics of the human gut microbiome in a case study of familial type 1 diabetes. Nat. Microbiol. 2: 16180. Erratum in Nat. Microbiol. 2: 16227.
H. Roume*, A. Heintz-Buschart*, E.E.L. Muller, P. May, V.P. Satagopam, C.C. Laczny, S. Narayanasamy, L.A. Lebrun, M.R. Hoopmann, J.M. Schupp, J.D. Gillece, N.D, Hicks, D.M. Engelthaler, T. Sauter, P.S. Keim, R.L. Moritz, P. Wilmes (2015). Comparative integrated omics: identification of key functionalities in microbial community-wide metabolic networks. NPJ Biofilms Microbiomes 1: 15007.
Code zu Publikationen
dadasnake Pipeline: https://github.com/a-h-b/dadasnake
Text mining und automatisierter Zugriff auf Sequenzdaten für Jurburg et al. (2020): https://github.com/a-h-b/pdf_to_sequence
R-script zu Goldmann et al., Environmental Microbiology (2019): https://a-h-b.github.io/SCALEMIC-betaDiv/
Repository zu Wampach, Heintz-Buschart und Fritz et al., Nature Communications (2018): https://git-r3lab.uni.lu/Cosmic/Earliest
Repository zu Heintz-Buschart et al., BMC Biology (2018): https://git.ufz.de/metaOmics/contaminomics
Repository zu Heintz-Buschart et al., Nature Microbiology (2016): https://git-r3lab.uni.lu/anna.buschart/MuStMultiomics
Lebenslauf
Heute arbeite ich als Assistant Professor in Mikrobieller Metagenomik am Swammerdam Institute for Life Sciences der Universität von Amsterdam.
September 2017 - Juli 2021
Wissenschaftliche Mitarbeiterin
Helmholtz Zentrum für Umweltforschung, Halle - Abteilung Bodenökologie
Deutsches Zentrum für integrative Biodiversitätsforschung (iDiv) Halle-Jena-Leipzig - Metagenomics Support Unit (2017-2020: Bioinformatics Unit)
Bioinformatik and Statistik für Metagenomik und funktionale (Meta-)omics:
- Methodenentwiklung für Multi-omics mikrobieller Gemeinschaften zur Analyse der Struktur und Funktion mikrobieller Gemeinschaften.
- Integration von physiologischen und Umweltparametern mit mikrobiellen Censusdaten.
2012 - 2017
Postdoc
Luxembourg Centre for Systems Biomedicine, Luxembourg - Arbeitsgruppe Eco-Systems Biology
Multi-omics-Analysen gemischter mikrobieller Gemeinschaften:
- statistische und ökologische Analysen der Struktur und Funktion mikrobieller Gemeinschaften im menschlichen Mikrobiom und im Belebtschlamm.
- bioinformatische Methodenentwicklung zur Integration von Multi-omics-Daten aus Next-Generation Sequenzierungen und Massenspektrometrie.
- Entwicklung und Leitung eines automatisierten Robotersystems zu Extraktion von Biomolekülen.
2011 - 2012
Postdoc
Helmholtz Zentrum für Infektionsforschung, Braunschweig - Projektgruppe Biologische Systemanalyse
Interaktion von Zellen des angeborenen Immunsystems mit Candida albicans:
- Modellierung und experimentelle Bewertung von Signaltransduktions- und Genregulationsnetzwerken.
- Analyse von Transkriptomdaten.
2008 - 2011
Helmholtz Zentrum für Infektionsforschung, Braunschweig - Projektgruppe Biologische Systemanalyse
Titel: "Wirkstoffsuche in Candida albicans: Etablierung neuer Screenings und Untersuchung der Wirkung antimykotischen Substanzen"
2002 - 2008
Diplom in Biologie
Universität Bremen
Hauptfach Mikrobiologie, Nebenfächer Botanik und Zellbiologie - Diplomarbeit: "Untersuchung der bakteriellen und pflanzlichen Genexpression in der Interaktion von Azoarcus sp. strain BH72 und Reis"
Publikationsliste (bis Juli 2021)
S.F.M. Wahdan, T. Reitz, A. Heintz-Buschart, M. Schädler, C. Roscher, C. Breitkreuz, B. Schnabel, W. Purahong, F. Buscot (2021): Organic agricultural practice enhances arbuscular mycorrhizal symbiosis in correspondence to soil warming and altered precipitation patterns. Environmental Microbiology and Environmental Microbiology Reports DOI: 10.1111/1462-2920.15492
R.M. Chakaroun, L. Massier, A. Heintz-Buschart, N. Said, J. Fallmann, A. Crane, T. Schütz, A. Dietrich, M. Blüher, M. Stumvoll, N. Musat, P. Kovacs (2021): Circulating bacterial signature is linked to metabolic disease and shifts with metabolic alleviation after bariatric surgery. Genome Medicine 13: 105.
C. Bueno de Mesquita, L. Nichols, M. Gebert, C. Vanderburgh, G. Bocksberger, J. Lester, A. Kalan, P. Dieguez, M. McCarthy, A. Agbor, P. Álvarez Varona, E. Ayimisin, M. Bessone, R. Chancellor, H. Cohen, C. Coupland, T. Deschner, V. Ebot Egbe, A. Goedmakers, A.-C. Granjon, C. Grueter, J. Head, R.A. Hernandez-Aguilar, K. Jeffery, S. Jones, P. Kadam, M. Kaiser, J. Lapuente, B. Larson, S. Marrocoli, D. Morgan, B. Mugerwa, F. Mulindahabi, E. Neil, P. Niyigaba, L. Pacheco, A. Piel, M. Robbins, A. Rundus, C. Sanz, L. Sciaky, D. Sheil, V. Sommer, F. Stewart, E. Ton, J. van Schijndel, V. Vergnes, E. Wessling, R. Wittig, Y. Ginath Yuh, K. Yurkiw, K. Zuberbühler, J. Gogarten, A. Heintz-Buschart, A. Muellner-Riehl, C. Boesch, H. Kühl, N. Fierer, M. Arandjelovic, R. Dunn (2021) Structure of the chimpanzee gut microbiome across their range. mSystems 6: e01269-20.
M. Ganther, D. Vetterlein, A. Heintz-Buschart, M.T. Tarkka (2021): Transcriptome sequencing analysis of maize roots reveals the effects of substrate and root hair formation in a spatial context. Plant and Soil DOI: 10.1007/s11104-021-04921-0 .
L. de Nies, S. Lopes, A. Heintz-Buschart, C.C. Laczny, P. May, P. Wilmes (2021): PathoFact: A pipeline for the prediction of virulence factors and antimicrobial resistance genes in metagenomic data. Microbiome 9:49.
S.B. Busi, L. de Nies, J. Habier, L. Wampach, J.V. Fritz, A. Heintz-Buschart, P. May, R. Halder, C. de Beaufort, P. Wilmes (2021): Persistence of birth mode-dependent effects on gut microbiome composition, immune system stimulation and antimicrobial resistance during the first year of life. ISME Communications 1:8.
J. Moll*, A. Heintz-Buschart*, C. Bässler, M. Hofrichter, H. Kellner, F. Buscot, B. Hoppe (2021): Amplicon sequencing-based bipartite network analysis confirms a high degree of specialization and modularity for fungi and prokaryotes in deadwood. mSphere, DOI: 10.1128/mSphere.00856-20.
S.F.M. Wahdan, A. Heintz-Buschart, C. Sansupa, B. Tanunchai, Y.-T. Wu, M. Schädler, M. Noll, W. Purahong, F. Buscot (2021). Targeting the active rhizosphere microbiome of Trifolium pratense in grassland evidences a stronger-than-expected belowground biodiversity-ecosystem functioning link. Frontiers in Microbiology 12: 73.
L. Gebauer, M.-L. Bouffaud, M. Ganther, B. Yim, D. Vetterlein, K. Smalla, F. Buscot, A. Heintz-Buschart, M.T. Tarkka (2021). Soil texture, sampling depth and root hairs shape the structure of ACC deaminase bacterial community composition in maize rhizosphere. Frontiers in Microbiology 12:128.
C. Weißbecker, B. Schnabel, A. Heintz-Buschart (2020): dadasnake, a Snakemake implementation of DADA2 to process amplicon sequencing data for microbial ecology. GigaScience 9: giaa135.
C.A. Guerra, A. Heintz-Buschart, J. Sikorski, A. Chatzinotas, N. Guerrero-Ramírez, S. Cesarz, L. Beaumelle, M.C. Rillig, F.T. Maestre, M. Delgado-Baquerizo, F. Buscot, J. Overmann, G. Patoine, H.R.P. Phillips, M. Winter, T. Wubet, K. Küsel, R.D. Bardgett, E.K. Cameron, D. Cowan, T. Grebenc, C. Marín, A. Orgiazzi, B.K. Singh, D.H. Wall, N. Eisenhauer (2020): Blind spots in global soil biodiversity and ecosystem function research. Nature Communications 11: 3870.
S.D. Jurburg, M. Konzack, N. Eisenhauer, A. Heintz-Buschart (2020): The archives are half-empty: an assessment of the availability of microbial community sequencing data. Communications Biology 3: 474.
L.D. Prada-Salcedo, K. Goldmann, A. Heintz-Buschart, T. Reitz, J. Wambsganss, J. Bauhus, F. Buscot (2020): Fungal guilds and soil functionality respond to tree community traits rather than to tree diversity in European forests. Molecular Ecology, https://doi.org/10.1111/mec.15749.
M. Widdig, A. Heintz-Buschart, P.M. Schleuss, A. Guhr, E.T. Borer, E.W. Seabloom, M. Spohn (2020): Effects of nitrogen and phosphorus addition on microbial community composition and element cycling in a grassland soil. Soil Biology and Biochemistry 151: 108041.
M. Herold, S. Martinez Arbas, S. Narayanasamy, A.R. Sheik, L.A.K. Kleine Borgmann, L. A. Lebrun, B. Kunath, H. Roume, I. Bessarab, R.B.H. Williams, J. Gillece, J. Schupp, P.S. Keim, C. Jäger, M. Hoopmann, R. Moritz, Y. Ye, S. Li, H. Tang, A. Heintz-Buschart, P. May, E.E.L. Muller, C.C. Laczny, P. Wilmes (2020): Integration of time-series meta-omics data reveals how microbial ecosystems respond to disturbance. Nature Communications 11, 5281.
F. Fischer, R. Romero, A. Hellhund, U. Linne, W. Bertrams, O. Pinkenburg, H. Shams Eldin, K. Binder, R. Jacob, A. Walker, B. Stecher, M. Basic, M. Luu, R. Mahdavi, A. Heintz-Buschart, A. Visekruna, U. Steinhoff (2020): Dietary cellulose induces anti-inflammatory immunity and transcriptional programs via maturation of the intestinal microbiota. Gut Microbes 12: 1-17.
M. Ganther, B. Yim, Z. Ibrahim, M.D. Bienert, E. Lippold, L. Maccario, S.J. Sørensen, G.P. Bienert, D. Vetterlein, A. Heintz-Buschart, E. Blagodatskaya, K. Smalla, M.T. Tarkka (2020): Compatibility of X-ray computed tomography with plant gene expression, rhizosphere bacterial communities and enzyme activities. Journal of Experimental Botany eraa262.
P.-M. Schleuss, M. Widdig, A. Heintz-Buschart, K. Kirkman, M. Spohn (2020): Interactions of nitrogen and phosphorus cycling promote P acquisition and explain synergistic plant growth responses. Ecology e03003.
K. Goldmann, R.S. Boeddinghaus, S. Klemmer, K.M. Regan, A. Heintz-Buschart, M. Fischer, D. Prati, H.-P. Piepho, D. Berner, S. Marhan, E. Kandeler, F. Buscot, T. Wubet (2020): Unraveling spatio-temporal variability of arbuscular mycorrhiza fungi in a temperate grassland plot. Environmental Microbiology 22(3): 873-888.
S. Cui, M. Li, R.Y.A. Hassan, A. Heintz-Buschart, J. Wang, U, Bilitewski (2020): Inhibition of Respiration of Candida albicans by Small Molecules Increases Phagocytosis Efficacy by Macrophages. mSphere 5: e00016-20.
C. Weißbecker, A. Heintz-Buschart, H. Bruelheide, F. Buscot, T. Wubet (2019): Linking Soil Fungal Generality to Tree Richness in Young Subtropical Chinese Forests. Microorganisms 7(11): 547.
O. Hickl, A. Heintz-Buschart, A. Trautwein-Schult, R. Hercog, P. Bork, P. Wilmes, D. Becher (2019): Sample preservation and storage significantly impact taxonomic and functional profiles in metaproteomics studies of the human gut microbiome. Microorganisms 7: 367.
P.-M. Schleuss, M. Widdig, A. Heintz-Buschart, A. Guhr, S. Martin, K. Kirkman, M. Spohn (2019): Stoichiometric controls of soil carbon and nitrogen cycling after long-term nitrogen and phosphorus addition in a mesic grassland in South Africa. Soil Biology & Biochemistry 135: 294-303.
A. Peters, P. Krumbholz, E. Jäger, A. Heintz-Buschart, M.V. Çakir, S. Rothemund, A. Gaudl, U. Ceglarek, T. Schöneberg, C. Stäubert (2019): Metabolites of lactic acid bacteria present in fermented foods are highly potent agonists of human hydroxycarboxylic acid receptor 3. PLoS Genetics 15: e1008145.
T.S.B. Schmidt, M.R. Hayward, L.P. Coelho, S.S. Li, P.I. Costea, A.Y. Voigt, J. Wirbel, O.M. Maistrenko, R.J. Alves, E. Bergsten, C. de Beaufort, I. Sobhani, A. Heintz-Buschart, S. Sunagawa, G. Zeller, P. Wilmes, P. Bork (2019): Extensive Transmission of Microbes along the Gastrointestinal Tract. eLife 8: e42693.
L. Wampach*, A. Heintz-Buschart*, J.V. Fritz*, J. Ramiro-Garcia, J. Habier, M. Herold, S. Narayanasamy, A. Kaysen, A.H. Hogan, L. Bindl, J. Bottu, R. Halder, C. Sjöqvist, P. May, A.F. Andersson, C. de Beaufort, P. Wilmes (2018): Birth mode is associated with earliest strain-conferred gut microbiome functions and immunostimulatory potential. Nature Communications 9: 5091.
A. Malabirade, J. Habier, A. Heintz-Buschart, P. May, J. Godet, R. Halder, A. Etheridge, D.J. Galas, P. Wilmes, J.V. Fritz (2018): The RNA Complement of Outer Membrane Vesicles From Salmonella enterica Serovar Typhimurium Under Distinct Culture Conditions. Frontiers in Microbiology 9: 2015.
A. Heintz-Buschart, D. Yusuf, A. Kaysen, A. Etheridge, J.V. Fritz, P. May, C. de Beaufort, B.B. Upadhyaya, A. Ghosal, D.J. Galas, P. Wilmes (2018): Small RNA profiling of low biomass samples: identification and removal of contaminants. BMC Biology 16: 52.
A.E. Laloo, J. Wei, D. Wang, S. Narayanasamy, I. Vanwonterghem, D.W. Waite, J. Steen, A. Kaysen, A. Heintz-Buschart, Q. Wang, B.L. Schulz, A.S. Nouwens, P. Wilmes, P. Hugenholtz, Z. Yuan, P.L. Bond (2018): Mechanisms of persistence of the ammonia oxidising bacteria- Nitrosomonas to the biocide Free Nitrous Acid (FNA). Environmental Science & Technology 52: 5386–97.
J. Habier, P. May, A. Heintz-Buschart, A. Ghosal, A.K. Wienecke-Baldacchino, E.N.M. Nolte-'t Hoen, P. Wilmes, J.V. Fritz (2018): Extraction and analysis of RNA isolated from pure bacteria-derived outer membrane vesicles. Methods in Molecular Biology 1737: 213-230.
A. Heintz-Buschart, U. Pandey, T. Wicke, A. Janzen, E. Sittig-Wiegand, C. Trenkwalder, W.H. Oertel, B. Mollenhauer, P. Wilmes (2017): The nasal and gastrointestinal microbiome in Parkinson’s disease and idiopathic REM sleep behavior disorder. Movement Disorders 6: e28032–11.
A. Kaysen, A. Heintz-Buschart, E.E.L. Muller, S. Narayanasamy, L. Wampach, C.C. Laczny, K. Franke, J. Bittenbring, J.G. Schneider, P. Wilmes (2017): Integrated meta-omic analyses of the gastrointestinal tract microbiome in patients undergoing allogeneic stem cell transplantation. Translational Research 186: 79-94.
L. Wampach, A. Heintz-Buschart, A. Hogan, E.E.L. Muller, S. Narayanasamy, C.C. Laczny, L.W. Hugerth, L. Bindl, J. Bottu, A.F. Andersson, C. de Beaufort, P. Wilmes (2017): The dynamics of archaea, bacteria and microeukaryotes within the human gastrointestinal microbiome during the first year of life according to birth mode. Frontiers in Microbiology 2: 738.
S. Narayanasamy, Y. Jarosz, E.E.L. Muller, A. Heintz-Buschart, M. Herold, A. Kaysen, C.C. Laczny, N. Pinel, P. May, P. Wilmes: IMP: a reproducible pipeline for reference-independent integrated metagenomic and metatranscriptomic analyses. Genome Biology 17: 260.
A. Heintz-Buschart, P. May, C.C. Laczny, L.A. Lebrun, C. Bellora, A. Krishna, L. Wampach, J.G. Schneider, A. Hogan, C. de Beaufort, P. Wilmes (2016): Integrated multi-omics of the human gut microbiome in a case study of familial type 1 diabetes. Nature Microbiology 2: 16180. Erratum in Nature Microbiology 2: 16227.
C.C. Laczny, E.E.L. Muller, A. Heintz-Buschart, M. Herold, L.A. Lebrun, A. Hogan, P. May, C. de Beaufort, P. Wilmes (2016): Identification, recovery, and refinement of hitherto undescribed population-level genomes from the human gastrointestinal tract. Frontiers in Microbiology 7: 884.
S. Cui, R.Y. Hassan, A. Heintz-Buschart, U. Bilitewski (2016): Regulation of Candida albicans interaction with macrophages through the activation of HOG pathway by genistein. Molecules 21: E162.
H. Roume*, A. Heintz-Buschart*, E.E.L. Muller, P. May, V.P. Satagopam, C.C. Laczny, S. Narayanasamy, L.A. Lebrun, M.R. Hoopmann, J.M. Schupp, J.D. Gillece, N.D, Hicks, D.M. Engelthaler, T. Sauter, P.S. Keim, R.L. Moritz, P. Wilmes (2015). Comparative integrated omics: identification of key functionalities in microbial community-wide metabolic networks. NPJ Biofilms Microbiomes 1: 15007.
X. Chen, L. Miché, S. Sachs, Q. Wang, A. Buschart, H. Yang, C. Vera Cruz, B. Reinhold-Hurek, T. Hurek, (2015): Rice responds to endophytic colonization which is independent of the common symbiotic signaling pathway. New Phytologist 208: 531-43.
C. Mathay, G. Hamot, E. Henry, L. Georges, C. Bellora, L. Lebrun, B. de Witt, W. Ammerlaan, A. Buschart, P. Wilmes, F. Betsou (2015): Method optimization for fecal sample collection and fecal DNA extraction. Biopreservation and Biobanking 13: 79-93.
A. Ghosal, B.B. Upadhyaya, J.V. Fritz, A. Heintz-Buschart, M.S. Desai, D. Yusuf, D. Huang, A. Baumuratov, K. Wang, D. Galas, P. Wilmes (2015): The extracellular RNA complement of Escherichia coli. Microbiologyopen doi: 10.1002/mbo3.235.
E.E.L. Muller, N. Pinel, C.C. Laczny, M.R. Hoopmann, S. Narayanasamy, L.A. Lebrun, H. Roume, J. Lin, P. May, N.D. Hicks, A. Heintz-Buschart, L. Wampach, C.M. Liu, L.B. Price, J.D. Gillece, C. Guignard, J.M. Schupp, N. Vlassis, N.S. Baliga, R.L. Moritz, P.S. Keim, P. Wilmes (2014): Community-integrated omics links dominance of a microbial generalist to fine-tuned resource usage. Nature Communication 26: 5603.
H. Roume, A. Heintz-Buschart, E.E.L. Muller, P. Wilmes (2013): Sequential isolation of metabolites, RNA, DNA, and proteins from the same unique sample. Methods in Enzymology 531: 219-36.
A. Heintz-Buschart, H. Eickhoff, E. Hohn, U. Bilitewski (2013): Identification of inhibitors of yeast-to-hyphae transition in Candida albicans by a reporter screening assay. Journal of Biotechnology 164: 137-42.
A. Buschart, A. Burakowska, U. Bilitewski (2012): The fungicide fludioxonil antagonizes fluconazole activity in the human fungal pathogen Candida albicans. Journal of Medical Microbiology 61: 1696-703.
A. Buschart*, K. Gremmer*, M. El-Mowafy, J. van den Heuvel, P.P. Müller, U. Bilitewski (2012): A novel functional assay for inhibitors of fungal histidine kinases type III reveals the role of HAMP domains for fungicide sensitivity. Journal of Biotechnology 157: 268-77.
A. Buschart*, S. Sachs*, X. Chen, J. Herglotz, A. Krause, B. Reinhold-Hurek (2012): Flagella mediate endophytic competence rather than act as MAMPS in rice-Azoarcus sp. strain BH72 interaction. Molecular Plant Microbe Interactions 25: 191-9.
M. Marselle, T. Hartig, D. Cox, S. de Bell, S. Knapp, S. Lindley, M. Triguero-Mas, K. Böhning-Gaese, P.A. Cook, S. de Vries, A. Heintz-Buschart, M. Hofmann, K.N. Irvine, N. Kabisch, F. Kolek, R. Kraemer, I. Markevych, D. Martens, R. Müller, M. Nieuwenhuijsen, J.M. Potts, J. Stadler, S. Walton, S.L. Warber, A. Bonn (2021): Pathways linking biodiversity to human health: a conceptual framework. Environment International, 150: 106420.
N. Eisenhauer, F. Buscot, A. Heintz-Buschart, S.D. Jurburg, K. Küsel, J. Sikorski, H.-J. Vogel, C.A. Guerra (2021): The multidimensionality of soil macroecology. Global Ecology and Biogeography 30: 4-10.
H.R.P. Phillips, A. Heintz-Buschart, N. Eisenhauer (2020): Putting soil invertebrate diversity on the map. Molecular Ecology 9: 655–657.
A. Heintz-Buschart, P. Wilmes (2017): Human Gut Microbiome: Function Matters. Trends in Microbiology 26: 563--74.
J.V. Fritz, A. Heintz-Buschart, A. Ghosal, L. Wampach, A. Etheridge, D. Galas, P. Wilmes (2016): Sources and functions of extracellular small RNAs in human circulation. Ann. Rev. Nutr. 36: 301--36.
P. Wilmes, A. Heintz-Buschart, P.L. Bond (2015): A decade of metaproteomics: where we stand and what the future holds. Proteomics 15: 3409--17.
