Das Mikrobiom und die metabolische Interaktion mit dem Wirt
Das intestinale Mikrobiom ist eine mikrobielle Gemeinschaft angesiedelt im Darmtrakt. Diese komplexe Gemeinschaft setzt sich hauptsächlich aus Bakterien zusammen und deckt mehrere hundert Spezies ab, wobei nur 50-100 verschiedene Bakterienarten in signifikanter Größe vorkommen. Die Zusammensetzung verändert sich nicht nur entlang der verschiedenen Darmabschnitte, sondern auch zwischen der Schleimhaut, die das Darmgewebe schützt und dem Darmlumen, welches Umwelteinflüssen ausgesetzt ist. Die komplexe Mikrobiota hat außerdem eine hohe metabolische und redundante funktionale Kapazität.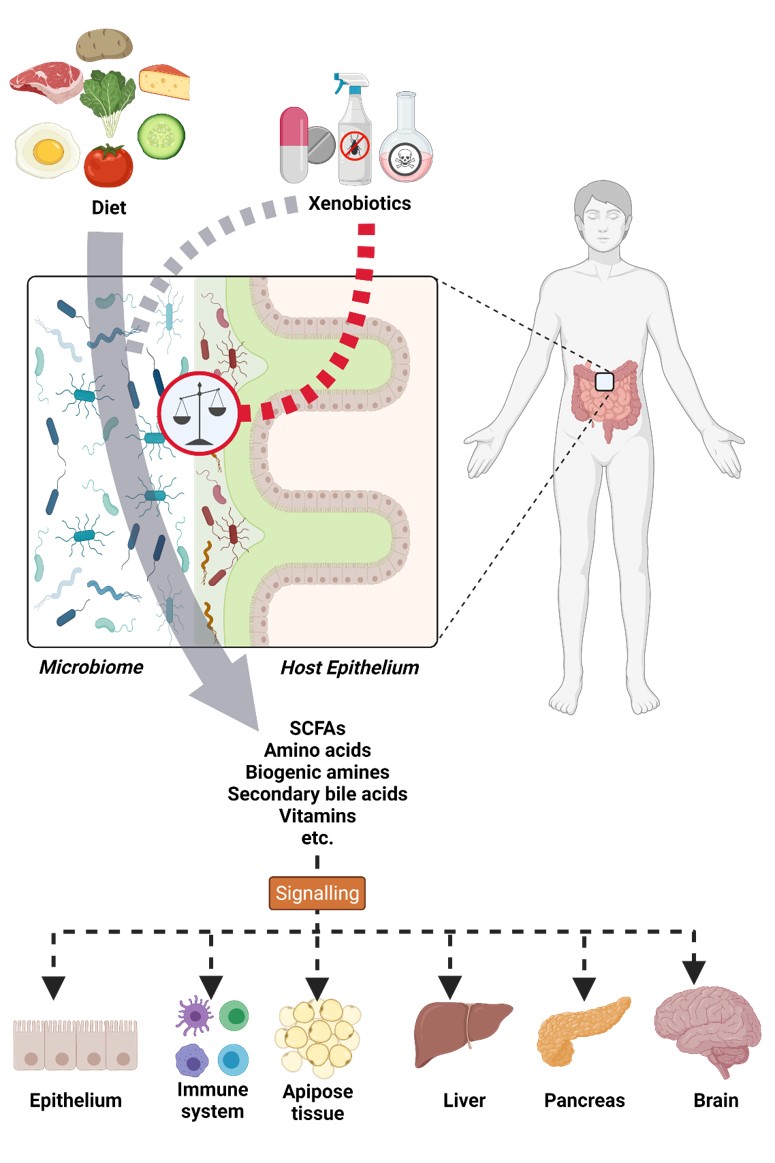
Das Mikrobiom erfüllt viele wichtige Funktionen für den Wirt. Darunter fällt die Umwandlung nicht verdaulicher Nahrungsbestandteile (zB. Kurzkettige Fettsäuren) sowie die Umwandlung ausgeschiedener Moleküle (zB. primäre Gallensäuren in sekundäre Gallensäuren) oder auch die komplette Neusynthese von Verbindungen (zB. Vitamine), die dann vom Wirt aufgenommen und verwertet werden.
All diese Metabolite stellen nicht nur eine Hauptnahrungsquelle dar sondern nehmen außerdem Schlüsselfunktionen in einer Mikrobiom-Wirts Interaktion ein. So können sie als Signalmoleküle in verschiedenen Organen des Wirtes fungieren und sind außerdem in der Lage das Immunsystem zu beeinflussen.
Die funktionellen Wechselwirkungen innerhalb des Mikrobioms sowie zwischen Mikrobiom und Wirt können durch externe Stressoren wie Xenobiotika (z.B. Medikamente, Pestizide und Umweltschadstoffe) verändert und gestört werden (genannt Dysbiose). Somit ist die Analyse dessen von entscheidender Bedeutung für das Verständnis der daraus resultierenden gesundheitlichen Folgen. Darüber hinaus können viele Xenobiotika vom Mikrobiom verstoffwechselt werden, bei denen wesentlich ist zu wissen, ob dies für den Wirt vorteilhaft oder nachteilig ist.
Um Antworten auf diese Problematiken zu finden, haben wir eine Vielzahl von analytischen Methoden etabliert. Diese decken 16S rRNA Gene Profiling, Metatranskriptomik, gerichtete und ungerichtete Metabolomik sowie metabolische Flussanalysen ab. Für die erweiterte Datenauswertung haben wir eine Vielzahl von Software Tools entwickelt, die die Datenanalyse und –integration automatisieren.
Wir messen sowohl Darm- und Kotproben von Menschen sowie aus Tierversuchsstudien.Zusammen mit der Arbeitsgruppe „Mikrobiombiologie“ verfügen wir außerdem über ein Chemostat-Bioreaktorsystem mit kontinuierlichem Fluss, welches mit verschiedenen Modellgemeinschaften von Darmmikrobiota betrieben werden kann.
All diese Metabolite stellen nicht nur eine Hauptnahrungsquelle dar sondern nehmen außerdem Schlüsselfunktionen in einer Mikrobiom-Wirts Interaktion ein. So können sie als Signalmoleküle in verschiedenen Organen des Wirtes fungieren und sind außerdem in der Lage das Immunsystem zu beeinflussen.
Die funktionellen Wechselwirkungen innerhalb des Mikrobioms sowie zwischen Mikrobiom und Wirt können durch externe Stressoren wie Xenobiotika (z.B. Medikamente, Pestizide und Umweltschadstoffe) verändert und gestört werden (genannt Dysbiose). Somit ist die Analyse dessen von entscheidender Bedeutung für das Verständnis der daraus resultierenden gesundheitlichen Folgen. Darüber hinaus können viele Xenobiotika vom Mikrobiom verstoffwechselt werden, bei denen wesentlich ist zu wissen, ob dies für den Wirt vorteilhaft oder nachteilig ist.
Um Antworten auf diese Problematiken zu finden, haben wir eine Vielzahl von analytischen Methoden etabliert. Diese decken 16S rRNA Gene Profiling, Metatranskriptomik, gerichtete und ungerichtete Metabolomik sowie metabolische Flussanalysen ab. Für die erweiterte Datenauswertung haben wir eine Vielzahl von Software Tools entwickelt, die die Datenanalyse und –integration automatisieren.
Wir messen sowohl Darm- und Kotproben von Menschen sowie aus Tierversuchsstudien.Zusammen mit der Arbeitsgruppe „Mikrobiombiologie“ verfügen wir außerdem über ein Chemostat-Bioreaktorsystem mit kontinuierlichem Fluss, welches mit verschiedenen Modellgemeinschaften von Darmmikrobiota betrieben werden kann.
