Ungerichtete Metabolom-Analyse mittels hochauflösender Massenspektrometrie
Metaboliten spiegeln sowohl intrinsische biologische als auch Umweltfaktoren wider. Es ist daher nicht verwunderlich, dass das Ziel, einen umfassenden Überblick über das gesamte Metabolom zu erhalten, das ultimative Ziel bleibt. Die ungerichtete Metabolomik ermöglicht die Sammlung von Daten ohne Vorwissen über die zu erwartenden Metaboliten und bietet somit auch die Möglichkeit, unbekannte Verbindungen zu detektieren. Eine gängige Methode zur Erzeugung von Daten in der ungezielten Metabolom-Analyse ist die Flüssigchromatographie in Verbindung mit der hochauflösenden Massenspektrometrie (Orbitrap oder Flugzeit-bestimmung als Massenanalysatoren).
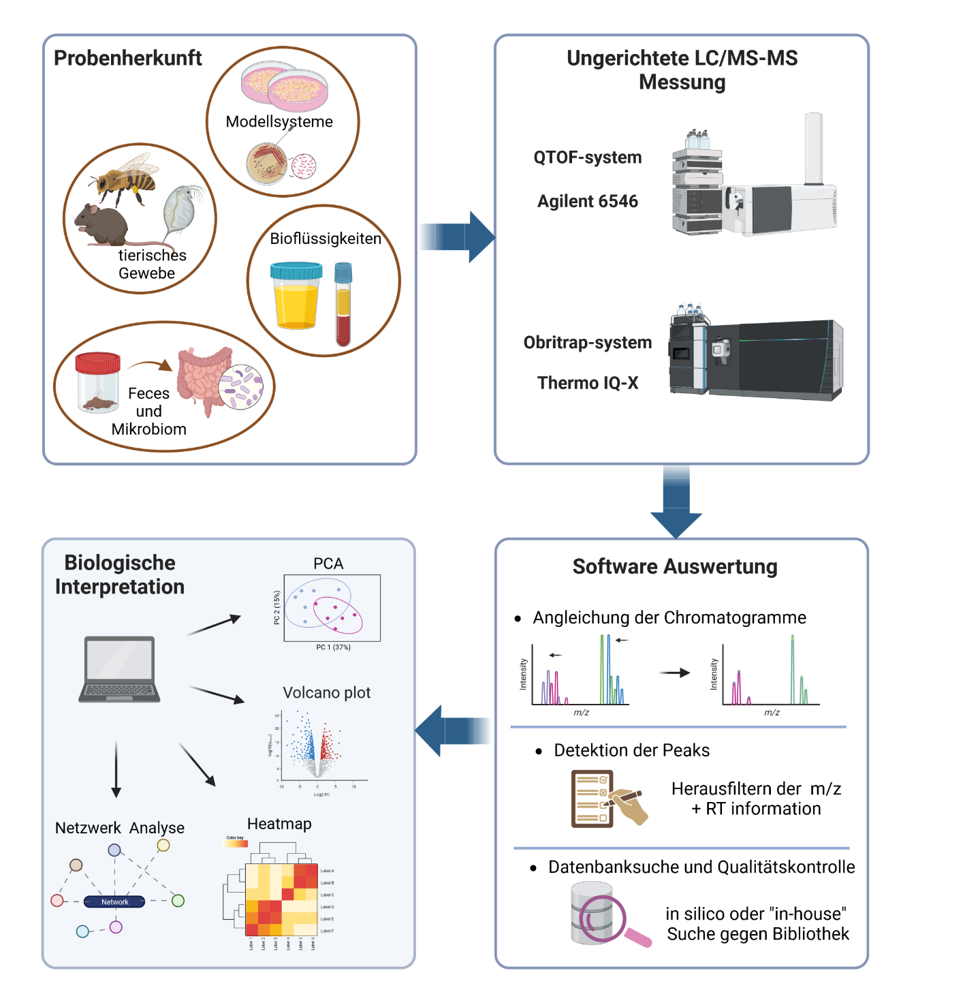
Das unvoreingenommene Screening zur Aufdeckung des gesamten Metaboloms kann auf eine Vielzahl von Proben angewendet werden, darunter Bioflüssigkeiten, Gewebeproben, Umweltproben und mikrobielle Proben. Die erhaltenen Daten stellen "Features" dar (Masse-Ladungs-Verhältnisse mit einer bestimmten Retentionszeit), die Metaboliten zugeordnet werden können. Leider stimmen die erhaltenen Informationen oft mit einer großen Anzahl möglicher Metabolitstrukturen überein oder es kann gar kein Kanditat mittels Datenbank-Suche identifiziet werden. Zur genauen Identifizierung spezifischer Feature müssen in einem zweiten Schritt Validierungsprozesse folgen. Da die biologische Interpretation auch von der Fähigkeit abhängt, Metaboliten valide zu identifizieren, bleibt dieser Prozess eine wichtige Herausforderung. Um diesem Problem zu begegnen, haben wir in unserer Arbeitsgruppe angefangen eine Metaboliten-Bibliothek zu implementieren. Dabei messen wir analytische Standards, die wir dann immer wieder in Retentionszeit und Masse-zu-Ladungs-Verhältnis plus Fragment-Muster mit unseren gemessenen Proben vergleichen können, um so die Validität der Identifizierung zu erhöhen.
