Metatranskriptomik – Funktionale Einblicke in mikrobielle Gemeinschaften
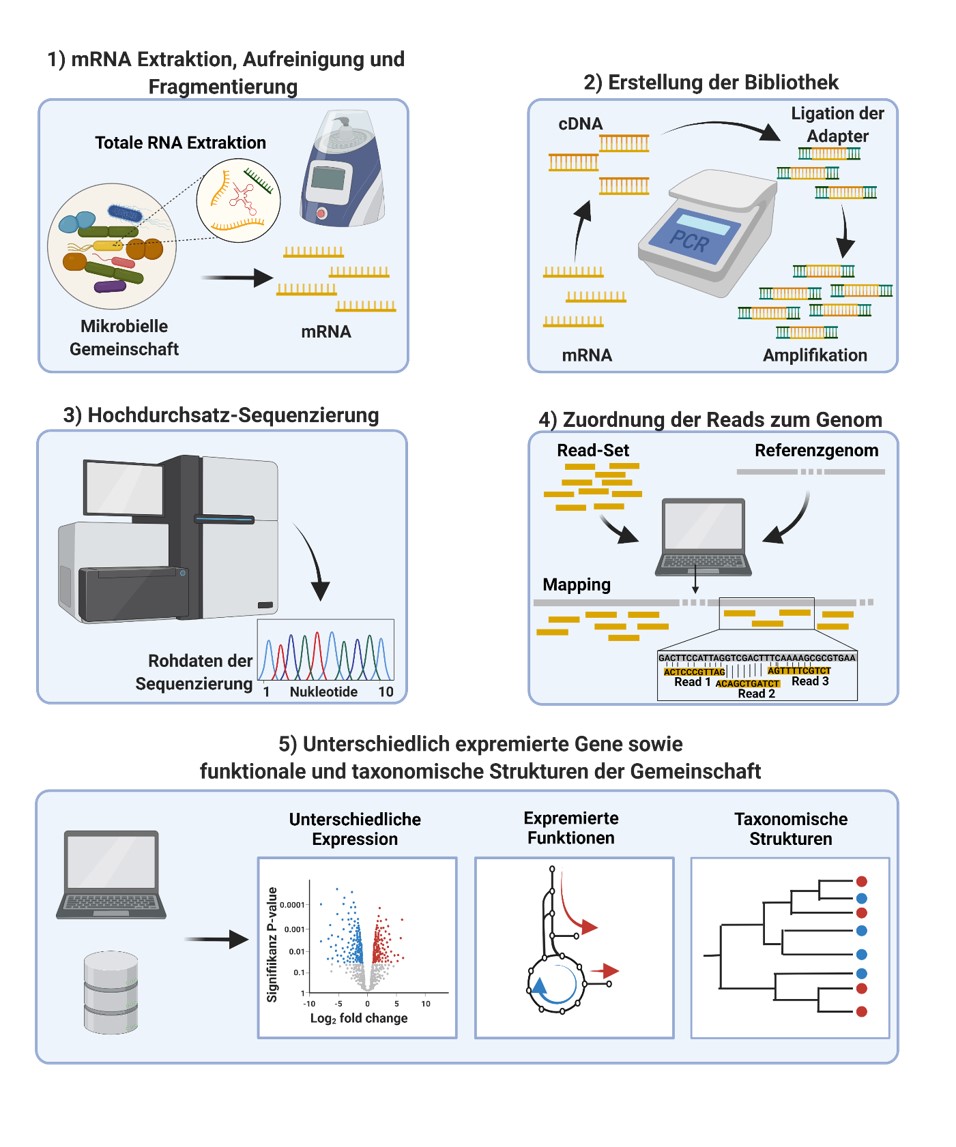
Nach der Probenahme und Extraktion der RNA aus einer Bakteriengemeinschaft wird die mRNA aufgereinigt und fragmentiert. Mittels reverser Transkription werden die mRNA-Fragmente in cDNA umgewandelt. Damit erfolgt die Erstellung einer Bibliothek, die die Ligation von Adaptern und die PCR-Amplifikation für das Next Generation Sequencing (NGS) umfasst. Nach der Sequenzierung werden die generierten „Reads“ mit einem zugehörigem Referenzgenom von bestimmten Bakterienstämmen abgeglichen. Die Anzahl der „Reads“ pro Gen ist ein Maß für die Häufigkeit des Transkripts in der untersuchten mikrobiellen Gemeinschaft. Diese Information kann dann zur Bestimmung der taxonomischen Struktur verwendet werden. Darüber hinaus können Transkripte mithilfe von Datenbanken (z. B. KEGG, EggNOG) funktionell annotiert werden und somit funktionellen Pfaden zugeordnet werden. So können mit metatranskriptomischen Analysen Veränderungen in der Taxonomie und in den Funktionen des Mikrobioms nachgewiesen werden. Diese Veränderungen können zu einer gestörten Mikrobiom-Wirts-Kommunikation führen und daher große Auswirkungen auf die Gesundheit haben.
In vielen experimentellen Studien verwenden wir ein kontinuierliches Kultivierungssystem (Bioreaktor), in welchem Bakterien mit Chemikalien oder anderen Stressoren exponiert werden können, um diese dann hinsichtlich ihrer Zusammensetzung und Funktion im Laufe der Zeit zu überwachen. Unsere Arbeitsgruppe hat eine bioinformatische Pipeline auf dem Galaxy-Server am Helmholtz-Zentrum für Umweltforschung - UFZ eingerichtet, um metatranskriptomische Sequenzierungsdaten zu analysieren.
