MADDOMS
Neue ultrahochauflösende massenspektrometrische Ansätze zur Analyse struktureller und physikochemischer Treiber der Stabilisierung von organischem Material

Schwerpunktprogramm 2322 ‘‘SoilSystems: Systemökologie von Böden – Energieaustrag reguliert durch Mikrobiom und Randbedingungen” gefördert durch die Deutsche Forschungsgemeinschaft (DFG, German Research Foundation). Link
Beteiligte Mitarbeiter der Abteilung Analytik am UFZ: Dr. Oliver Lechtenfeld (PI), Dr. Carsten Simon (Postdoc), Konstantin Stumpf (Doktorand), Prof. Dr. Thorsten Reemtsma (Abteilungsleiter)
Externe Beteiligte: Dr. Marion Schrumpf (Bodenbiogeochemie, Max Planck Institut für Biogeochemie, Jena), Prof. Dr. Robert Mikutta Dr. Klaus Kaiser (beide Bodenkunde und Bodenschutz, Martin Luther University Halle-Wittenberg)
Überblick. MADDOMS ist ein Projekt im Rahmen des Schwerpunktprogramms 2322 ''SoilSystems: Systemökologie von Böden", das von der Deutschen Forschungsgemeinschaft (DFG) gefördert wird. In diesem SPP wird der Boden als ein hochkomplexes, offenes thermodynamisches System betrachtet, in dem Struktur, Funktion und Stabilität des Bodenökosystems von Energieabgabe und - verbrauch abhängen. Organische Substanz und mikrobielle Biomasse in Böden können in diesem Rahmen als dissipative Strukturen betrachtet werden. Die drei Haupthypothesen des SPP sind:
1) Das Bodenmikrobiom ist ein wichtiger Regler der Energiedissipation und des Stoffumsatzes (es trägt über Metabolismus, Recycling und Nekromasse-Akkumulation zur Bildung der organischen Bodensubstanz, engl. soil organic matter (SOM) bei) und folgt dabei spezifischen Energienutzungswegen.
2) Die Zufuhr von Energie und Materie, ihre Abfuhr und ihr Verbrauch prägen die biologische Komplexität der Böden, z. B. die Vielfalt des Mikrobioms oder die Organisation des Nahrungsnetzes.
3) Die Randbedingungen (z. B. Sauerstoff- oder Nährstoffverfügbarkeit) und die Mineralogie bestimmen die Kanäle, die für die Energie- und Stoffnutzung zur Verfügung stehen.
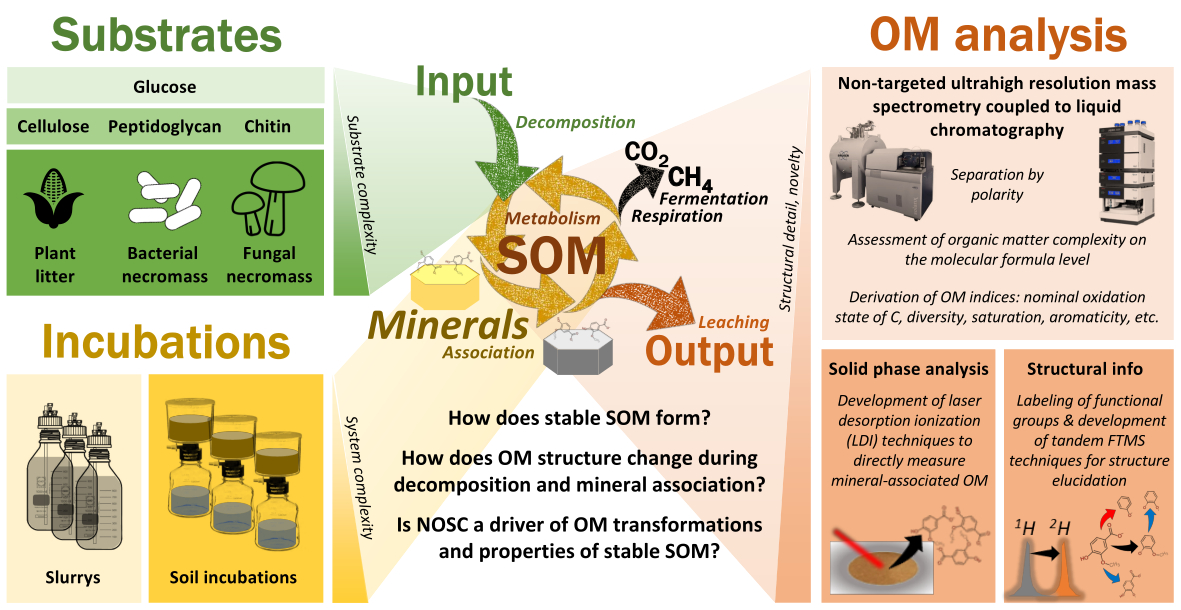
Zielsetzung. MADDOMS kombiniert Inkubationsversuche mit der Entwicklung neuartiger Analysewerkzeuge zur Bewertung der SOM-Bildung und der Veränderungen in der molekularen Zusammensetzung (Fig 1). Das Projekt zielt darauf ab, neue Anwendungen von Informationen auf molekularer Ebene durch ultrahochauflösende Massenspektrometrie in der Bodenkunde zu etablieren: strukturelle Eigenschaften (über Tandem-MS und Isotopenmarkierungsansätze) und oberflächenassoziierte Moleküle (über Laserdesorptionstechniken, engl. laser desorption ionization, LDI).
Der nominelle Oxidationszustand (engl. nominal oxidation state of carbon, NOSC) als Indikator für die Stabilität von SOM. Flüssigchromatographie gekoppelt mit ultrahochauflösender Massenspektrometrie ermöglicht den Zugang zur metabolischen Signatur des Abbaus von organischem Material [1]. Diese „Signaturen“ umfassen Eigenschaften wie das Molekulargewicht, den Sättigungs- und Oxidationsgrad, die Polarität, die Aromatizität und den Energiegehalt (NOSC). Die energetischen Eigenschaften von SOM sind nicht gut erforscht, weshalb eine zentrale Frage der SOM-Persistenz bisher ungelöst ist - warum entstehen bei der SOM-Bildung energetisch günstige Produkte, die offensichtlich nicht verbraucht, sondern angereichert werden [2]? MADDOMS wird zu einem besseren Verständnis der thermodynamischen Grundlagen der SOM-Bildung durch neue Analysewerkzeuge beitragen, die den Energie- und Stoffumsatz in Böden bewerten (Hypothese 1).
Die organische Substanz als Spiegel der funktionalen Komplexität des Bodens. Böden sind offene, mehrphasige, heterogene und lebende Systeme, die häufigen Veränderungen der Randbedingungen unterliegen (z.B. Trockenheit, Zufuhr an organischem Material, etc.) [3, 4]. Diese Systeme sind durch parallele und miteinander verknüpfte Prozesse gekennzeichnet, die zur Bildung hochkomplexer Gemische von organischen Molekülen führen [5,6]. MADDOMS wird zu einem besseren Verständnis der funktionellen Komplexität von SOM beitragen und dazu, wie diese auf die Art des Ausgangssubstrats (Pflanze – Pilz – Bakterium), den Abbau, die Redox-Bedingungen (liegt Sauerstoff vor oder nicht) und die Exposition gegenüber mineralischen Oberflächen reagiert. Die funktionelle Komplexität wird dabei nicht nur auf der Ebene der Summenformel [7], sondern auch auf der strukturellen Ebene [8-10] betrachtet. Eine weitere Ebene zum Verständnis der funktionellen Komplexität wird durch den Vergleich zwischen gelösten und mineral-assoziierten (sorbierten) Molekülen geschaffen (Hypothese 2) [11,12].
Grenzbedingungen steuern die Bildungswege und die Zusammensetzung der stabilen organischen Substanz. Die Stabilität oder Persistenz der organischen Bodensubstanz wird heute als eine Eigenschaft des Ökosystems und nicht nur als eine Frage der unterschiedlichen Abbaubarkeit bestimmter Strukturen oder Biomaterialien betrachtet („Rekalzitranz“) [13-15]. Im Rahmen des SPP werden Inkubationsversuche die Möglichkeit bieten, beide Effekte auf der Grundlage von drei großen Substratkategorien (Pflanze - Pilz - Bakterium) zu testen. MADDOMS wird die Auswirkungen des Substratabbaus auf die molekularen und strukturellen Eigenschaften der erzeugten organischen Substanz untersuchen und sie mit den Signaturen von natürlichem SOM vergleichen. Langfristige Inkubationen werden Einblicke in die Abhängigkeit dieser Prozesse von externen (Randbedingungen) und internen Faktoren (biochemisches Potenzial des Mikrobioms) liefern (Hypothese 3).
Weiterführende Literatur
[1] Han, L.; Kaesler, J.; Peng, C.; Reemtsma, T.; Lechtenfeld, O. J. (2021): Anal. Chem. 93: 1740. Link
[2] Gunina, A.; Kuzyakov, Y. (2022): Glob. Chang. Biol. 28: 2169. Link
[3] Addiscott, T. (2010): Geoderma. 160: 31. Link
[4] Sierra, C. A.; Müller, M.; Metzler, H.; Manzoni, S.; Trumbore, S. E. (2017): Glob. Chang. Biol. 23: 1763. Link
[5] Lehmann, J.; Hansel, C. M.; Kaiser, C.; Kleber, M.; Maher, K.; Manzoni, S.; Nunan, N.; Reichstein, M.; Schimel, J. P.; Torn, M. S.; Wieder, W. R.; Kögel-Knabner, I. (2020): Nat. Geosci. 13: 529. Link
[6] Lehmann, J.; Kleber, M. (2015): Nature 528: 60. Link
[7] Mentges, A.; Feenders, C.; Seibt, M.; Blasius, B.; Dittmar, T. (2017): Front. Mar. Sci. 4: 194. Link
[8] Kostyukevich, Y.; Kononikhin, A.; Zherebker, A.; Popov, I.; Perminova, I.; Nikolaev, E. (2014): Anal. Bioanal. Chem. 406: 6655. Link
[9] Zherebker, A.; Kostyukevich, Y.; Kononikhin, A.; Kharybin, O.; Konstantinov, A. I.; Zaitsev, K. V.; Nikolaev, E.; Perminova, I. (2017): Anal. Bioanal. Chem. 409: 2477. Link
[10] Simon, C.; Dührkop, K.; Petras, D.; Roth, V.-N.; Böcker, S.; Dorrestein, P. C.; Gleixner, G. (2022): Environ. Sci. Technol. 56: 11027. Link
[11] Lohse, M.; Haag, R.; Lippold, E.; Vetterlein, D.; Reemtsma, T.; Lechtenfeld, O. J. (2021): Front. Plant Sci. 12: 753812. Link
[12] Giannopoulos, K.; Benettoni, P.; Holbrook, T. R.; Reemtsma, T.; Wagner, S.; Lechtenfeld, O. J. (2021): Environ. Sci. Nano 8: 2336. Link
[13] Marschner, B.; Brodowski, S.; Dreves, A.; Gleixner, G.; Gude, A.; Grootes, P. M.; Hamer, U.; Heim, A.; Jandl, G.; Ji, R.; Kaiser, K.; Kalbitz, K.; Kramer, C.; Leinweber, P.; Rethemeyer, J.; Schäffer, A.; Schmidt, M. W. I.; Schwark, L.; Wiesenberg, G. L. B. (2008): J. Plant Nutr. Soil Sci. 171: 91. Link
[14] Schmidt, M. W. I.; Torn, M. S.; Abiven, S.; Dittmar, T.; Guggenberger, G.; Janssens, I. A.; Kleber, M.; Kögel-Knabner, I.; Lehmann, J.; Manning, D. A. C.; Nannipieri, P.; Rasse, D. P.; Weiner, S.; Trumbore, S. E. (2011): Nature 478: 49. Link
[15] Roth, V.-N.; Lange, M.; Simon, C.; Hertkorn, N.; Bucher, S.; Goodall, T.; Griffiths, R. I.; Mellado-Vázquez, P. G.; Mommer, L.; Oram, N. J.; Weigelt, A.; Dittmar, T.; Gleixner, G. (2019): Nat. Geosci. 12: 755. Link
