Mikrobielle Interaktionsökologie
Unsere Arbeitsgruppe und das UFZ begrüßen Bewerbungen, Studierende und Mitarbeiter:innen unabhängig von Nationalität, Religion, Geschlechtsidentifikation, sexueller Orientierung, Alter oder Behinderung. Wir glauben an vielfältige Perspektiven und Erfahrungen und wollen so ein Umfeld schaffen, das hilft, ein breites Spektrum an möglichen Lösungen für wissenschaftliche Fragen, aber auch für die Gesellschaft im Allgemeinen zu finden.
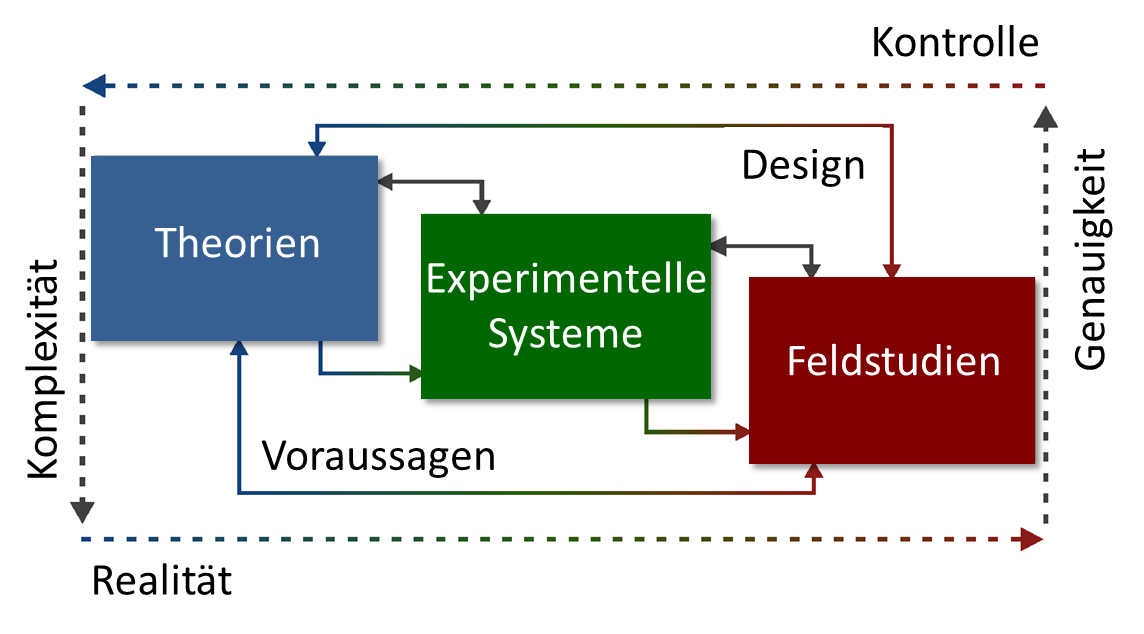
Forschungsinteressen
Natürliche und künstliche Ökosysteme bestehen aus mehreren trophischen Ebenen, die miteinander in Wechselwirkung stehen.
Während Bakterien die Hauptakteure vieler Funktionen sind, kontrollieren Viren und andere Mikroprädatoren (Protisten, bakterielle Räuber) diese Gemeinschaften und damit ihre Funktionen auf mehreren Ebenen. So hat die Infektion mit Viren nicht nur Auswirkungen auf die Zusammensetzung der Gemeinschaften und die genetische Landschaft, sondern auch auf den biogeochemischen Umsatz und das Recycling von Nährstoffen (z.B. viraler Shunt).
Unser Ziel ist es, die Diversität von Viren und Mikroben, ihre genomische Zusammensetzung, ihr funktionelles Potential und ihre Interaktionen sowie die sich daraus ergebenden Konsequenzen für Ökosysteme durch Theorie, bioinformatische Analysen und Data Mining, Laborexperimente und Feldstudien zu verstehen.Mit diesem Wissen wollen wir Lösungen für das Management von (mikrobiellen) Ökosystemen und die Anwendung von Viren/Prädatoren/mikrobiellen Inokula in natürlichen, biotechnologischen oder wirtsassoziierten Systemen anbieten.
Ein weiteres Anliegen ist das Management und die Förderung einer fairen gemeinsamen Datennutzung, um die Entdeckung neuer Erkenntnisse in der Mikrobiomforschung zu beschleunigen
Aktuelle Projekte und Kooperationen
- "Viraler shunt", Virome, und Viren-Bakterien/Viren-Pilz Interaktionen im terrestrischen Untergrund (mit DFG-CRC AquaDiva Partnern, AquaDiva, UFZ AG Bioverfügbarkeit, Lukas Wick)
- Virome in Böden unter verschiedenen Landnutzungssystemen (mit Michael Schloter, TU München, Helmholtz Zentrum München; Christian Griebler, Universität Wien; Biodiversitäts-Exploratorien SPP 1374)
- Mikrobiome in Böden unter verschiedenen Landnutzungs- und Klimaszenarien, und in der Rhizosphäre von Pflanzen/Bäumen; Biodiversitäts-Exploratorien SPP 1374), Phytoakmeter GCEF
- Auswirkungen von Pestiziden auf mikrobielle Nahrungsnetze im Boden (mit Dimitris Karpouzas, Universität Thessalien, Griechenland, EU-ITN ARISTO)
- Mikrobielle Bio-Pestizide und Risikobewertung für neue risikoarme Pestizide (low-risk pesticides), EU RATION
- Mikrobiome von Seen und Amphibien (mit Vance Vredenburg , San Francisco State University; ENSAT, Toulouse France; P3)
- Funktionale Merkmale von Phytoplankton entlang von Umweltgradienten (mit iDiv: Susanne Dunker, Stan Harpole, Adam Clark (Universität Graz)
- SARS-CoV-2 RNA im Abwasser (mit TU Dresden)
Wissenschaftler/-innen
Dr. Stephanie Jurburg: Mikrobielle Ökologie, Community Assembly, Data Stewardship
Dr. Nawras Ghanem: Viren-Bakterien-Interaktionen
Dr. João Pedro Leonor Fernandes Saraiva: Bakterielle (Meta)Genomik, Protistengenome, Biopestizide
Dr. Esteban Nieto: Protisten-Bakterien Interaktionen, mikrobielle Inokula
Felipe Borim Corrêa: Virale Metagenomik
Doktoranden/-innen
Marta Eugenia Perez Villanueva (Gast; gemeinsame Betreuung mit Cédric Malandain, HYDREKA/Lyon) : Effekte von Pestiziden auf mikrobielle Interaktions - und Nahrungsnetze ARISTO Projekt)
Peter Hofmann (gemeinsame Betreuung mit Susanne Dunker, Stan Harpole): Variabilität physiologischer und morphologische Merkmale von Phytoplankton entlang von Umweltgradienten, Mikrokosmenversuche
Maria Kostakou: Räumliche Zusammensetzung der mikrobieller Bodengemeinschaften in Abhängigkeit von räumlichen Skalen und Landnutzung Biodiversitätsexploratorien
Jiaming Ma
Lu Wang: Phytoakmeter https://www.uni-marburg.de/en/fb17/phytoakmeter/phytoakmeter-subprojecs/project-area-d
Techniker/-innen
Anett Heidtmann
Nicole Steinbach
Lara Zenker (Auszubildende)
Studenten/-innen
Cassidy Funke: Isolierung und Charaterisierung von preädatorischen Bakterien (Bdellovibrio) aus Umweltproben
Vanessa Erxleben: Bakterielle Lyse durch Viren und "Viral shunt" im Grundwasser
Gastwissenschaftler/-innen
-
Weitere Informationen über laufende Projekte und Kooperationen finden Sie auf den Internetseiten des Gruppenleiters Antonis Chatzinotas und der Gruppenmitglieder
Publikationen
Unsere Publikationen finden Sie auf den jeweiligen Internetseiten der Gruppenmitglieder.
Aktuell ausgeschriebene Postdoc- und Doktorandenstellen, sowie Master- Bachelorarbeiten und Praktika finden sich unter
Jobs und Ausbildung
