Forschungsthemen der Arbeitsgruppe "Toxicoproteomics"
Krankhaftes Übergewicht (Adipositas) stellt bei Kindern, Jugendlichen und Erwachsenen den größten Risikofaktor für die Entstehung von Stoffwechselerkrankungen wie z.B. Diabetes, Herzkreislauferkrankungen, Bluthochdruck, Fettleber und einige Krebsarten dar. Dabei konnten Metabolismus-disruptive Chemikalien (MDCs) mit der Entstehung von Adipositas und Begleiterkrankungen in Verbindung gebracht werden. Die Aufklärung der zugrunde liegenden pathophysiologischen Mechanismen ist essentiell um präventive Maßnahmen und effektive Behandlungsmethoden für Patienten mit Adipositas zu entwickeln.
Im Fettgewebe kann vor allem die Funktion von Adipozyten durch MDCs beeinflusst werden. Da Adipositas mit einer gering-gradigen Entzündung des Fettgewebes einhergeht, welche vor allem durch Makrophagen verursacht ist, sollten Makrophagen für die Charakterisierung von MDC-vermittelten Effekten als essentielle Akteure betrachtet werden. Wie MDCs die Interaktion beider Zelltypen beeinflussen, ist jedoch noch nicht geklärt.
Unsere Projekte zielen darauf ab, ein mechanistisches Verständnis der Auswirkungen von MDCs auf die Adipogenese und auf das Zusammenspiel von Adipozyten und Makrophagen sowie deren Funktionen zu erlangen. Mit Hilfe innovativer systembiologischer omics-Methoden, wie Bulk- und Einzelzellproteomik, thermal proteome profiling oder der Analyse von post-translationalen Modifikationen, werden molekulare Initiationsereignisse und Schlüsselereignisse identifiziert, welche Daten für die Entwicklung eines adverse outcome pathways (AOPs) liefern. Neben der Entschlüsselung der Wirkungsweise von MDCs definieren wir mit unsrer Forschung molekulare Signaturen welche die Auswirkungen von MDCs erklären und Biomarker für humanes Biomonitoring liefern.
Folgende Forschungsthemen werden bearbeitet:
Einfluss von Schadstoffen in Makrophagen
In Makrophagen, den wichtigsten Zellen des angeborenen Immunsystems, untersuchen wir den Einfluss von Schadstoffen wie Nanomaterialien, Mikro- und Nanoplastik, Weichmacher, Koservierungsstoffe oder polyzyklischen aromatischen Kohlenwasserstoffen im akuten Entzündungsgeschehen.
Dabei nutzen wir, neben klassischen molekularbiologischen Methoden, globale Proteomik in Kombination mit der Analyse von post-translationalen Modifkationen (Phosphorylierung, Acetylierung und Oxidierung).
Informationen: Stefanie Raps
Einfluss von Schadstoffen in Adipozyten
In früheren Arbeiten konnten wir zeigen, dass die Adipogenese und die Funktion von Adipozyten durch Umweltchemikalien, wie Weichmacher, verändert wird. Um die molekulare Wirkmechanismen von solchen Umweltchemikalien besser zu verstehen, nutzen wir, neben klassischen molekularbiologischen Methoden, globale Proteomik in Kombination mit der Analyse von post-translationalen Modifkationen (Phosphorylierung, Acetylierung und Oxidierung).
Post-translationale Modifikationen (PTMs)
Im Rahmen des DFG Sonderforschungsbereichs 1052 „Mechanismen der Adipositas“ nutzen wir Massenspektrometrie-basierte Methoden zur Charakterisierung von post-translationalen Modifkationen (Acetylierung und Phosphorylierung) während der Adipogenese und in reifen Adipozyten. Ein weiteres Ziel unserer Arbeiten ist es, die Beeinflussung von PTMs durch Umweltchemikalien und sich daraus ergebende funktionelle Konsequenzen zu verstehen.
Informationen:
Alix S. Aldehoff
SFB 1052
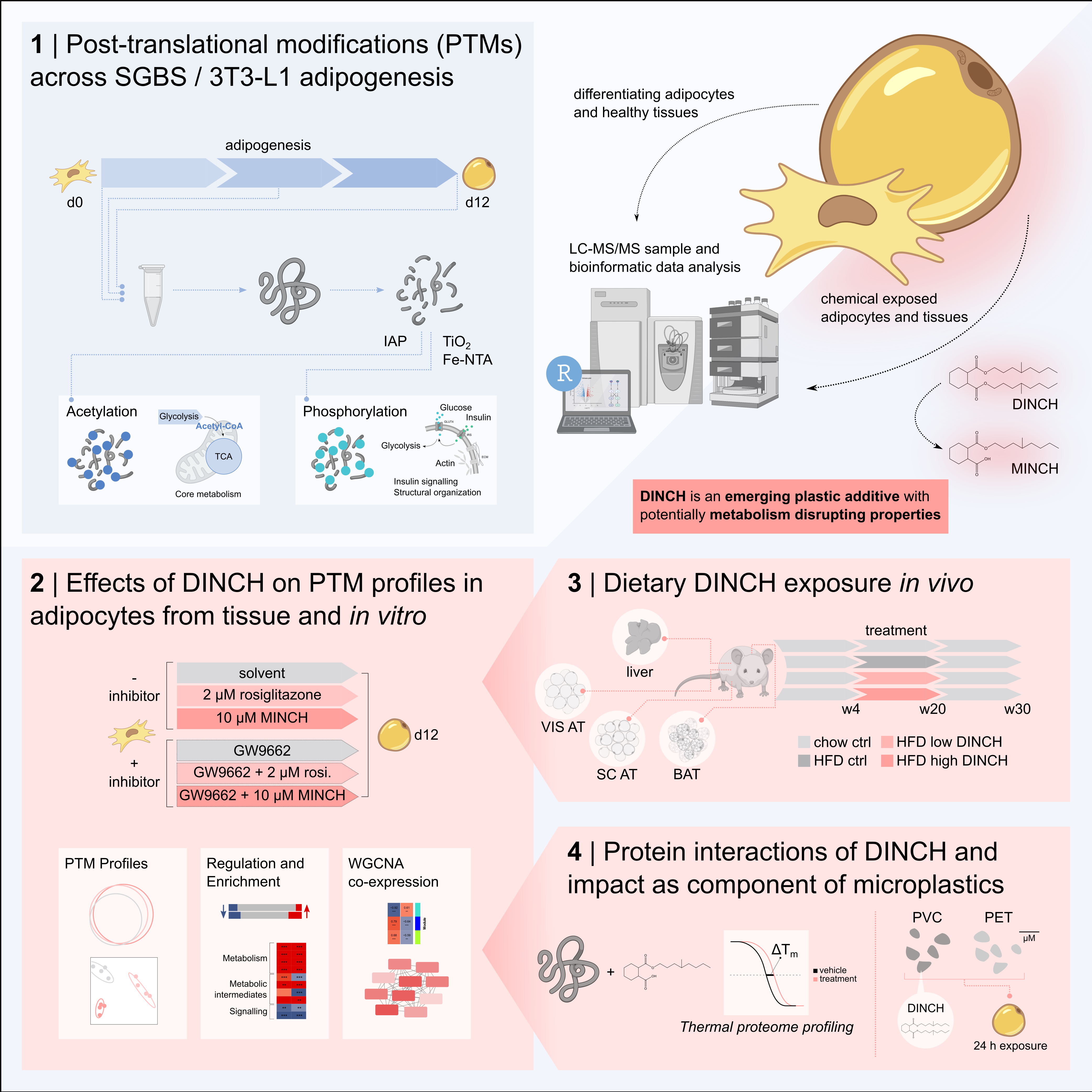
Adipogenese als neuer Endpunkt in der Risikobewertung
Im Rahmen der European Partnership for the Risk Assessment of Chemicals (
PARC
), etablieren wir eine Novel Approach Method für Adipogenese als Endpunkt in der Risikobewertung. Molekulare Effekte und zelluläre Zielproteine von bisher wenig untersuchten Bisphenol A Alternativen werden mit globaler Proteomik und Thermal Proteome Profiling charakterisiert.
Informationen: Jasmin Kleißen
Inhalt:
Weiterführende Recherchen können Sie in unserem Publikationsverzeichnis durchführen.
2024 (3)
- Aldehoff, A.S., Karkossa, I., Goerdeler, C., Krieg, L., Schor, J., Engelmann, B., Wabitsch, M., Landgraf, K., Hackermüller, J., Körner, A., Rolle-Kampczyk, U., Schubert, K., von Bergen, M. (2024):
Unveiling the dynamics of acetylation and phosphorylation in SGBS and 3T3-L1 adipogenesis
iScience , art. 109711 10.1016/j.isci.2024.109711 - Goerdeler, C., Engelmann, B., Aldehoff, A.S., Schaffert, A., Blüher, M., Heiker, J.T., Wabitsch, M., Schubert, K., Rolle-Kampczyk, U., von Bergen, M. (2024):
Metabolomics in human SGBS cells as new approach method for studying adipogenic effects: Analysis of the effects of DINCH and MINCH on central carbon metabolism
Environ. Res. 252 , art. 118847 10.1016/j.envres.2024.118847 - Raps, S., Bahr, L., Karkossa, I., Rossol, M., von Bergen, M., Schubert, K. (2024):
Triclosan and its alternatives, especially chlorhexidine, modulate macrophage immune response with distinct modes of action
Sci. Total Environ. 941 , art. 169650 10.1016/j.scitotenv.2023.169650
2023 (11)
- Aldehoff, A.S., Karkossa, I., Engelmann, B., Rolle-Kampczyk, U., von Bergen, M., Schubert, K. (2023):
Exploring the regulatory effects of lysine acetylation and phosphorylation in adipocyte differentiation and their exposure to emerging plastic additives
Toxicol. Lett. 384 (Suppl. 1), S133 - S133 10.1016/S0378-4274(23)00578-7 - Bifari, F., Dolci, S., Mannino, L., Campanelli, A., Bottani, E., Karkossa, I., Setten, E., Gianoli, S., Di Chio, M., Ciarpella, F., Busetto, G., Pezzotta, A., Castagna, A., Pruonto, G., Binaschi, L., Amenta, A., Scupoli, M., Cavallini, C., Boschi, F., Doherty, A., Malpeli, G., Rossi, E., Martinelli, N., Ferretti, S., Malik, Z., Sandri, M., Pistocchi, A., Schubert, K., Endrizzi, M., Bossolasco, P., Fumagalli, G., Locati, M., Decimo, I. (2023):
Adoptive transfer of tumor-educated macrophages promote neural tissue regeneration and motor recovery following severe spinal cord injury
Cytotherapy 25 (6, Suppl.), E8 - E9 10.1016/S1465-3249(23)00711-9 - Braeuning, A., Balaguer, P., Bourguet, W., Carreras-Puigvert, J., Feiertag, K., Kamstra, J.H., Knapen, D., Lichtenstein, D., Marx-Stoelting, P., Rietdijk, J., Schubert, K., Spjuth, O., Stinckens, E., Thedieck, K., van den Boom, R., Vergauwen, L., von Bergen, M., Wewer, N., Zalko, D. (2023):
Development of new approach methods for the identification and characterization of endocrine metabolic disruptors—a PARC project
Front. Toxicol. 5 , art. 1212509 10.3389/ftox.2023.1212509 - Gerhards, M., Böhme, A., Schubert, K., Kodritsch, B., Ulrich, N. (2023):
DNA adducts as link between in vitro and in vivo carcinogenicity – A case study with benzo[a]pyrene
Curr. Res. Toxicol. 4 , art. 100097 10.1016/j.crtox.2022.100097 - Gurbuz, B., Guldiken, N., Reuken, P., Fu, L., Remih, K., Preisinger, C., Brůha, R., Leníček, M., Petrtýl, J., Reissing, J., Aly, M., Fromme, M., Zhou, B., Karkossa, I., Schubert, K., von Bergen, M., Stallmach, A., Bruns, T., Strnad, P. (2023):
Biomarkers of hepatocellular synthesis in patients with decompensated cirrhosis
Hepatol. Int. 17 (3), 698 - 708 10.1007/s12072-022-10473-x - Höper, T., Karkossa, I., Dumit, V.I., von Bergen, M., Schubert, K., Haase, A. (2023):
A comparative proteomics analysis of four contact allergens in THP-1 cells shows distinct alterations in key metabolic pathways
Toxicol. Appl. Pharmacol. 475 , art. 116650 10.1016/j.taap.2023.116650 - Karkossa, I., Fürst, S., Großkopf, H., von Bergen, M., Schubert, K. (2023):
Oxidation is an underappreciated post-translational modification in the regulation of immune responses associated with changes in phosphorylation
Front. Immunol. 14 , art. 1244431 10.3389/fimmu.2023.1244431 - Lipaeva, P., Karkossa, I., Bedulina, D., Schubert, K., Luckenbach, T. (2023):
Cold-adapted amphipod species upon heat stress: Proteomic responses and their correlation with transcriptomic responses
Comp. Biochem. Physiol. D-Genomics Proteomics 45 , art. 101048 10.1016/j.cbd.2022.101048 - Remih, K., Hufnagel, F., Durkalski-Mauldin, V., Lee, W.M., Su, C., Rule, J., Krieg, L., Karkossa, I., Schubert, K., von Bergen, M., Fontana, R., Strnad, P. (2023):
Serum proteomics in American adults with acute liver failure
J. Hepatol. 78 (Suppl. 1), S136 - S138 10.1016/S0168-8278(23)00638-4 - Remih, K., Hufnagel, F.-M., Durkalski-Mauldin, V., Lee, W., Su, Z., Rule, J., Krieg, L., Karkossa, I., Schubert, K., von Bergen, M., Fontana, R., Strnad, P. (2023):
Serum proteomics in adults with Acute Liver Failure
Z. Gastroent. 61 (08), e460 - e461 10.1055/s-0043-1771831 - Wang, Z., Haange, S.-B., Haake, V., Huisinga, M., Kamp, H., Buesen, R., Schubert, K., Canzler, S., Hackermüller, J., Rolle-Kampczyk, U., von Bergen, M. (2023):
Assessing the influence of propylthiouracil and phenytoin on the metabolomes of the thyroid, liver, and plasma in a rats
Metabolites 13 (7), art. 847 10.3390/metabo13070847
2022 (5)
- Kage, P., Schubert, K., Treudler, R., Simon, J.-C., von Bergen, M., Tomm, J. (2022):
Identification of new potential allergens from green-lipped mussel (Perna canaliculus)
Iran. J. Allergy Asthma Immunol. 21 (6), 711 - 715 10.18502/ijaai.v21i6.11532 - Krieg, L., Didt, K., Karkossa, I., Bernhart, S.H., Kehr, S., Subramanian, N., Lindhorst, A., Schaudinn, A., Tabei, S., Keller, M., Stumvoll, M., Dietrich, A., von Bergen, M., Stadler, P.F., Laurencikiene, J., Krüger, M., Blüher, M., Gericke, M., Schubert, K., Kovacs, P., Chakaroun, R., Massier, L. (2022):
Multiomics reveal unique signatures of human epiploic adipose tissue related to systemic insulin resistance
Gut 71 (11), 2179 - 2193 10.1136/gutjnl-2021-324603 - Lentz, L.S., Stutz, A.J., Meyer, N., Schubert, K., Karkossa, I., von Bergen, M., Zenclussen, A.C., Schumacher, A. (2022):
Human chorionic gonadotropin promotes murine Treg cells and restricts pregnancy-harmful proinflammatory Th17 responses
Front. Immunol. 13 , art. 989247 10.3389/fimmu.2022.989247 - Murthy, S., Karkossa, I., Schmidt, C., Hoffmann, A., Hagemann, T., Rothe, K., Seifert, O., Anderegg, U., von Bergen, M., Schubert, K., Rossol, M. (2022):
Danger signal extracellular calcium initiates differentiation of monocytes into SPP1/osteopontin-producing macrophages
Cell Death Dis. 13 , art. 53 10.1038/s41419-022-04507-3 - Schaffert, A., Karkossa, I., Ueberham, E., Schlichting, R., Walter, K., Arnold, J., Blüher, M., Heiker, J.T., Lehmann, J., Wabitsch, M., Escher, B.I., von Bergen, M., Schubert, K. (2022):
Di-(2-ethylhexyl) phthalate substitutes accelerate human adipogenesis through PPARγ activation and cause oxidative stress and impaired metabolic homeostasis in mature adipocytes
Environ. Int. 164 , art. 107279 10.1016/j.envint.2022.107279
2021 (14)
- Großkopf, H., Vogel, S., Müller, C.D., Köhling, S., Dürig, J.-N., Möller, S., Schnabelrauch, M., Rademann, J., Hempel, U., von Bergen, M., Schubert, K. (2021):
Identification of intracellular glycosaminoglycan-interacting proteins by affinity purification mass spectrometry
Biol. Chem. 402 (11), 1427 - 1440 10.1515/hsz-2021-0167 - Großkopf, H., Walter, K., Karkossa, I., von Bergen, M., Schubert, K. (2021):
Non-genomic AhR-signaling modulates the immune response in endotoxin-activated macrophages after activation by the environmental stressor BaP
Front. Immunol. 12 , art. 620270 10.3389/fimmu.2021.620270 - Höper, T., Siewert, K., Dumit, V.I., von Bergen, M., Schubert, K., Haase, A. (2021):
The contact allergen NiSO4 triggers a distinct molecular response in primary human dendritic cells compared to bacterial LPS
Front. Immunol. 12 , art. 644700 10.3389/fimmu.2021.644700 - Karkossa, I., Bannuscher, A., Hellack, B., Wohlleben, W., Laloy, J., Stan, M.S., Dinischiotu, A., Wiemann, M., Luch, A., Haase, A., von Bergen, M., Schubert, K. (2021):
Omics reveal different oxidative stress levels induced by nanomaterials in vitro and in vivo
Toxicol. Lett. 350 (Suppl.), S125 10.1016/S0378-4274(21)00541-5 - Karkossa, I., Bannuscher, A., Hellack, B., Wohlleben, W., Laloy, J., Stan, M.S., Dinischiotu, A., Wiemann, M., Luch, A., Haase, A., von Bergen, M., Schubert, K. (2021):
Nanomaterials induce different levels of oxidative stress, depending on the used model system: Comparison of in vitro and in vivo effects
Sci. Total Environ. 801 , art. 149538 10.1016/j.scitotenv.2021.149538 - Monikh, F.A., Chupani, L., Karkossa, I., Gardian, Z., Arenas-Logo, D., von Bergen, M., Schubert, K., Piackova, V., Zuskova, E., Jiskoot, W., Vijver, M.G., Peijnenburg, W.J.G.M. (2021):
An environmental ecocorona influences the formation and evolution of the biological corona on the surface of single-walled carbon nanotubes
NanoImpact 22 , art. 100315 10.1016/j.impact.2021.100315 - Nickel, S., Vlaic, S., Christ, M., Schubert, K., Henschler, R., Tautenhahn, F., Burger, C., Kühne, H., Erler, S., Roth, A., Wild, C., Brach, J., Hammad, S., Gittel, C., Baunack, M., Lange, U., Broschewitz, J., Stock, P., Metelmann, I., Bartels, M., Pietsch, U.-C., Krämer, S., Eichfeld, U., von Bergen, M., Dooley, S., Tautenhahn, H.-M., Christ, B. (2021):
Mesenchymal stromal cells mitigate liver damage after extended resection in the pig by modulating thrombospondin-1/TGF-β
npj Regen. Med. 6 , art. 84 10.1038/s41536-021-00194-4 - Schaffert, A., Arnold, J., Karkossa, I., Blüher, M., von Bergen, M., Schubert, K. (2021):
The emerging plasticizer alternative DINCH and its metabolite MINCH induce oxidative stress and enhance inflammatory responses in human THP-1 macrophages
Cells 10 (9), art. 2367 10.3390/cells10092367 - Schaffert, A., Krieg, L., Weiner, J., Schlichting, R., Ueberham, E., Karkossa, I., Bauer, M., Landgraf, K., Junge, K.M., Wabitsch, M., Lehmann, J., Escher, B.I., Zenclussen, A.C., Körner, A., Blüher, M., Heiker, J.T., von Bergen, M., Schubert, K. (2021):
Alternatives for the worse: Molecular insights into adverse effects of bisphenol a and substitutes during human adipocyte differentiation
Environ. Int. 156 , art. 106730 10.1016/j.envint.2021.106730 - Schaffert, A., Krieg, L., Weinert, J., Schlichting, R., Ueberham, E., Karkossa, I., Bauer, M., Junge, K., Wabitsch, M., Lehmann, J., Escher, B.I., Zenclussen, A.C., Körner, A., Blüher, M., Heiker, J.T., von Bergen, M., Schubert, K. (2021):
Emerging substitutes of BPA cause inflammation-like state and impaired insulin sensitivity in differentiating human adipocytes
Toxicol. Lett. 350 (Suppl.), S130 10.1016/S0378-4274(21)00552-X - Schubert, K., Karkossa, I., Schor, J., Engelmann, B., Steinheuer, L.M., Bruns, T., Rolle-Kampczyk, U., Hackermüller, J., von Bergen, M. (2021):
A multi-omics analysis of mucosal-associated-invariant T cells reveals key drivers of distinct modes of activation
Front. Immunol. 12 , art. 616967 10.3389/fimmu.2021.616967 - Streidl, T., Karkossa, I., Segura Muñoz, R.R., Eberl, C., Zaufel, A., Plagge, J., Schmaltz, R., Schubert, K., Basic, M., Schneider, K.M., Afify, M., Trautwein, C., Tolba, R., Stecher, B., Doden, H.L., Ridlon, J.M., Ecker, J., Moustafa, T., von Bergen, M., Ramer-Tait, A.E., Clavel, T. (2021):
The gut bacterium Extibacter muris produces secondary bile acids and influences liver physiology in gnotobiotic mice
Gut Microbes 13 (1), 1 - 21 10.1080/19490976.2020.1854008 - Walter, K., Grosskopf, H., Karkossa, I., von Bergen, M., Schubert, K. (2021):
Proteomic characterization of the cellular effects of AhR activation by microbial tryptophan catabolites in endotoxin-activated human macrophages
Int. J. Environ. Res. Public Health 18 (19), 10336 10.3390/ijerph181910336 - Wang, Z., Karkossa, I., Großkopf, H., Canzler, S., Rolle-Kampczyk, U., Hackermüller, J., von Bergen, M., Schubert, K. (2021):
Quantitative proteomics reveal mechanistic insights into direct and indirect drug-induced thyroid toxicity in rats
Toxicol. Lett. 350 (Suppl.), S129 10.1016/S0378-4274(21)00550-6
2020 (9)
- Bannuscher, A., Karkossa, I., Buhs, S., Nollau, P., Kettler, K., Balas, M., Dinischiotu, A., Hellack, B., Wiemann, M., Luch, A., von Bergen, M., Haase, A., Schubert, K. (2020):
A multi-omics approach reveals mechanisms of nanomaterial toxicity and structure–activity relationships in alveolar macrophages
Nanotoxicology 14 (2), 181 - 195 10.1080/17435390.2019.1684592 - Canzler, S., Schor, J., Busch, W., Schubert, K., Rolle-Kampczyk, U.E., Seitz, H., Kamp, H., von Bergen, M., Buesen, R., Hackermüller, J. (2020):
Prospects and challenges of multi‑omics data integration in toxicology
Arch. Toxicol. 94 (2), 371 - 388 10.1007/s00204-020-02656-y - Hsu, M.-J., Karkossa, I., Schäfer, I., Christ, M., Kühne, H., Schubert, K., Rolle-Kampczyk, U.E., Kalkhof, S., Nickel, S., Seibel, P., von Bergen, M., Christ, B. (2020):
Mitochondrial transfer by human mesenchymal stromal cells ameliorates hepatocyte lipid load in a mouse model of NASH
Biomedicines 8 (9), art. 350 10.3390/biomedicines8090350 - Kalkhof, S., Krieg, L., Büttner, P., Wabitsch, M., Küntzel, C., Friebe, D., Landgraf, K., Hanschkow, M., Schubert, K., Kiess, W., Krohn, K., Blüher, M., von Bergen, M., Körner, A. (2020):
In depth quantitative proteomic and transcriptomic characterization of human adipocyte differentiation using the SGBS cell line
Proteomics 20 (15-16), art. 1900405 10.1002/pmic.201900405 - Karkossa, I., Raps, S., von Bergen, M., Schubert, K. (2020):
Systematic review of multi-omics approaches to investigate toxicological effects in macrophages
Int. J. Mol. Sci. 21 (24), art. 9371 10.3390/ijms21249371 - Krause, J.L., Haange, S.-B., Schäpe, S.S., Engelmann, B., Rolle-Kampczyk, U., Fritz-Wallace, K., Wang, Z., Jehmlich, N., Türkowsky, D., Schubert, K., Pöppe, J., Bote, K., Rösler, U., Herberth, G., von Bergen, M. (2020):
The glyphosate formulation Roundup® LB plus influences the global metabolome of pig gut microbiota in vitro
Sci. Total Environ. 745 , art. 140932 10.1016/j.scitotenv.2020.140932 - Krieg, L., Schaffert, A., Kern, M., Landgraf, K., Wabitsch, M., Beck-Sickinger, A.G., Koerner, A., Blüher, M., von Bergen, M., Schubert, K. (2020):
An MRM-based multiplexed quantification assay for human adipokines and apolipoproteins
Molecules 25 (4), art. 775 10.3390/molecules25040775 - Rolle-Kampczyk, U., Gebauer, S., Haange, S.-B., Schubert, K., Kern, M., Moulla, Y., Dietrich, A., Schön, M.R., Klöting, N., von Bergen, M., Blüher, M. (2020):
Accumulation of distinct persistent organic pollutants is associated with adipose tissue inflammation
Sci. Total Environ. 748 , art. 142458 10.1016/j.scitotenv.2020.142458 - Wang, Z., Karkossa, I., Großkopf, H., Rolle-Kampczyk, U., Hackermüller, J., von Bergen, M., Schubert, K. (2020):
Comparison of quantitation methods in proteomics to define relevant toxicological information on AhR activation of HepG2 cells by BaP
Toxicology 448 , art. 152652 10.1016/j.tox.2020.152652
2019 (7)
- Großkopf, H., Schubert, K., von Bergen, M. (2019):
AhR-dependent non-genomic signaling in macrophages - A multi-PTM-omics and proteomics study
Eur. J. Immunol. 49 (Suppl. 1), 137 10.1002/eji.201970300 - Karkossa, I., Bannuscher, A., Hellack, B., Bahl, A., Buhs, S., Nollau, P., Luch, A., Schubert, K., von Bergen, M., Haase, A. (2019):
An in-depth multi-omics analysis in RLE-6TN rat alveolar epithelial cells allows for nanomaterial categorization
Part. Fibre Toxicol. 16 (1), art. 38 10.1186/s12989-019-0321-5 - Kratochvil, I., Bannuscher, A., Hellack, B., von Bergen, M., Haase, A., Schubert, K. (2019):
Nanomaterials affect oxidative phosphorylation and phagocytosis in alveolar macrophages
Eur. J. Immunol. 49 (Suppl. 1), 232 10.1002/eji.201970300 - Schmidt, J.R., Geurtzen, K., von Bergen, M., Schubert, K., Knopf, F. (2019):
Glucocorticoid treatment leads to aberrant ion and macromolecular transport in regenerating zebrafish fins
Front. Endocrinol. 10 , art. 674 10.3389/fendo.2019.00674 - Schmidt, J.R., Vogel, S., Moeller, S., Kalkhof, S., Schubert, K., von Bergen, M., Hempel, U. (2019):
Sulfated hyaluronic acid and dexamethasone possess a synergistic potential in the differentiation of osteoblasts from human bone marrow stromal cells
J. Cell. Biochem. 120 (5), 8706 - 8722 10.1002/jcb.28158 - Schubert, K., Collins, L.E., Green, P., Nagase, H., Troeberg, L. (2019):
LRP1 controls TNF release via the TIMP-3/ADAM17 axis in endotoxin-activated macrophages
J. Immunol. 202 (4), 1501 - 1509 10.4049/jimmunol.1800834 - Schubert, K., Schor, J., Kratochvil, I., Hackermüller, J., von Bergen, M. (2019):
Multi-omics profiling of MAIT cells reveals specific patterns of antigen-dependent and -independent activation
Eur. J. Immunol. 49 (Suppl. 1), 141 10.1002/eji.201970300
2018 (2)
- Firacative, C., Gressler, A.E., Schubert, K., Schulze, B., Müller, U., Brombacher, F., von Bergen, M., Alber, G. (2018):
Identification of T helper (Th)1- and Th2-associated antigens of Cryptococcus neoformans in a murine model of pulmonary infection
Sci. Rep. 8 , art. 2681 10.1038/s41598-018-21039-z - Schmidt, J.R., Rücker-Braun, E., Heidrich, K., von Bonin, M., Stölzel, F., Thiede, C., Middeke, J.M., Ehninger, G., Bornhäuser, M., Schetelig, J., Schubert, K., von Bergen, M., Heidenreich, F. (2018):
Pilot study on mass spectrometry–based analysis of the proteome of CD34+CD123+ progenitor cells for the identification of potential targets for immunotherapy in acute myeloid leukemia
Proteomes 6 (1), art. 11 10.3390/proteomes6010011
2017 (1)
- Schubert, K., Olde Damink, S.W.M., von Bergen, M., Schaap, F.G. (2017):
Interactions between bile salts, gut microbiota, and hepatic innate immunity
Immunol. Rev. 279 (1), 23 - 35 10.1111/imr.12579
Inhalt:
Weiterführende Recherchen können Sie in unserem Publikationsverzeichnis durchführen.
2024 (1)
- Raps, S., Schubert, K. (2024):
Effects of Triclosan and its alternatives on LPS-stimulated THP-1 macrophages
ProteomeXchange 10.6019/PXD044828
2023 (3)
- Schubert, K., Karkossa, I. (2023):
Effects of high molecular weight phthalates on LPS-stimulated THP-1 macrophages - proteome
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD043025 - Schubert, K., Karkossa, I. (2023):
Effects of high molecular weight phthalates on LPS-stimulated THP-1 macrophages - redoxome
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD043139 - Schubert, K., Karkossa, I. (2023):
Effects of high molecular weight phthalates on LPS-stimulated THP-1 macrophages - phosphoproteome
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD042912
2022 (4)
- Schubert, K., Karkossa, I. (2022):
Proteomic characterization of calcium macrophages
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD026888 - Schubert, K., Schaffert, A. (2022):
Effects of plasticizers on adult human adipocytes (SGBS) using global proteomics
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD030843 - Schubert, K., Schaffert, A. (2022):
THP-1 cells treated with plasticizer DINCH and metabolite MINCH
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD027744 - Schubert, K., Schaffert, A. (2022):
Effects of plasticizers on human preadipocytes (SGBS) using global proteomics
PRoteomics IDEntifications Database (PRIDE) 10.6019/PXD031041
proteomicsr: An analysis pipeline for label-based and label-free proteomics data
Creator: Karkossa, Isabel
Description
This package summarizes helpful functions to analyze global information on protein abundance (proteomics) and the abundance of post-translational modifications (PTMs), referred to as PTM-omics. Thereby, it supports data from label-free and label-based approaches. For this purpose, functions for initial data evaluation and clean-up, data transformation, determination of significantly affected candidates, and different typically presented proteomics plots are provided. Furthermore, it contains functions to perform enrichment analyses as well as to visualize significantly enriched terms as well as candidates assigned to such terms. Enrichment analyses are performed based on gene sets of the MSigDB, but also the visualization of results obtained using the Ingenuity Pathway Analysis (IPA, Qiagen) tool is possible. Last but not least, the steps necessary to perform a Weighted Gene Correlation Network Analysis (WGCNA) are included, starting with the network creation and finally exporting potential key drivers of the effects (so-called hub genes).
Although this package has been established for proteomics data, it works (in parts) for other omics layers as well. For instance, the determination of significantly altered candidates can be applied to metabolomics data, the enrichment using MSigDB gene sets can be applied to transcriptomics data, and the WGCNA is applicable to all omics layers, even for integrative data evaluation.
The current version is available on Zenodo or Git:
zenodo
git
