Protein stable isotope probing (Protein-SIP)
Für die Charakterisierung von mikrobiellen Lebensgemeinschaften wurde in Kooperation mit dem Department Isotopenbiogeochemie das Protein-SIP-Verfahren (SIP = „stable isotope probing“) entwickelt, dass die gleichzeitige Bestimmung der Spezies und ihrer metabolischen Aktivität ermöglicht. So ist es möglich, mikrobielle Gemeinschaften auf taxonomischer und funktioneller Ebene zu untersuchen.
Die Assimilation von schweren, stabilen Isotopen in die Biomoleküle verschiedener Spezies ist abhängig vom Umsatz und der metabolischen Aktivität der Spezies. Der Einbau stabiler Isotope durch ein Substrat kann zum Nachweis der metabolisch-aktiven Spezies innerhalb einer Lebensgemeinschaft genutzt werden. Die verschiedenen Einbaustufen werden durch die Intensität der Blaufärbung verdeutlicht.
Nach der Zellernte und Proteinextraktion werden die Proben proteolytisch behandelt und mittels Massenspektrometrie analysiert. Die Isotopologen werden, je nach Einbau schwerer Isotope in die Proteine, in einem höheren Massenbereich detektiert. Ein höherer Einbaugrad steht für eine stärkere Assimilation und demzufolge für eine primäre Rolle der Spezies bei der Nutzung des markierten Substrates innerhalb des Nahrungsnetzes. Der Einbau schwerer, stabiler Isotope in die Peptide / Proteine kann zum Beispiel durch die Anwendung der ‚Halben Dezimalstellen Regel’ abgeschätzt werden. Wir haben in Zusammenarbeit mit der Universität Tübingen eine bioinformatische Lösung entwickelt (MetaProSIP), die eine Quantifizierung des metabolischen Einbaus in Proteine ermöglicht.
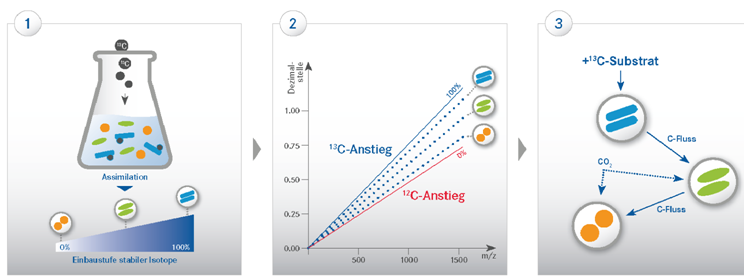
Peptide ermöglichen eine phylogenetische Information zur strukturellen Aufklärung der Lebensgemeinschaft und zur physiologischen Beschreibung des aktuellen Zustandes der mikrobiellen Zellen. Anhand dieser Informationen können Kohlenstoffflüsse und Nahrungsnetzwerke aufgeklärt werden und sie helfen im Weiteren die Interaktionen innerhalb der mikrobiellen Lebensgemeinschaften zu verstehen.
